Cientistas que estudam uma enzima de coronavírus COVID-19 em temperaturas que variam de geladas a quentes do corpo humano descobriram mudanças estruturais sutis que oferecem pistas sobre como a enzima funciona. As descobertas, publicadas no IUCrJ, o jornal da União Internacional de Cristalografia, podem inspirar o design de novos medicamentos para combater o COVID-19 – e possivelmente ajudar a evitar futuras pandemias de coronavírus.
“Nenhum estudo anterior analisou essa importante enzima do coronavírus à temperatura fisiológica (ou corporal)”, disse Daniel Keedy, biólogo estrutural da Universidade da Cidade de Nova York (CUNY), que conduziu o estudo em colaboração com cientistas do Departamento dos EUA. do Laboratório Nacional Brookhaven da Energia.
A maioria das estruturas até hoje vem de amostras congeladas – longe das temperaturas nas quais as moléculas operam dentro das células vivas. “Se você está trabalhando em temperatura fisiológica, deve ter uma imagem mais realista do que está acontecendo durante uma infecção real, porque é aí que a biologia acontece”, disse Keedy.
Além disso, acrescentou, a equipe usou a temperatura como ferramenta. “Ao girar esse botão e ver como a proteína reage, podemos aprender sobre sua mecânica – como ela funciona fisicamente.
Decifrando a estrutura do Mpro
A proteína em questão é a principal protease (Mpro) do SARS-CoV-2, o vírus que causa o COVID-19. Como todas as proteases, é uma enzima que corta outras proteínas. Em muitas infecções virais, incluindo COVID-19, as células infectadas inicialmente produzem proteínas funcionais de um vírus como uma única cadeia de proteínas conectada. As proteases separam os pedaços para que as proteínas individuais possam produzir e se montar em novas cópias do vírus. Encontrar um medicamento para desativar o Mpro pode frear o COVID-19.
Para estudar a estrutura da enzima, os pesquisadores usaram uma técnica chamada cristalografia de raios-X na National Synchrotron Light Source II (NSLS-II) do Brookhaven Lab. O NSLS-II é uma instalação do usuário do DOE Office of Science que produz feixes brilhantes de raios-x. O brilho desses raios X em uma amostra cristalizada de uma molécula biológica pode revelar o arranjo tridimensional dos átomos que compõem a molécula.
Estudar amostras que não estão congeladas pode ser um desafio.
“Quanto mais alta a temperatura, maiores as chances de os raios X danificarem o cristal”, explicou o coautor do estudo Babak Andi, que opera a linha de luz Frontier Macromolecular Crystallography (FMX) do NSLS-II.
“Para minimizar os danos, giramos e movemos o cristal linearmente à medida que ele se move através dos raios X. Isso distribui a dose de raios X por todo o comprimento do cristal”, disse ele.
Ele observou que o pequeno tamanho do feixe de raios X no NSLS-II torna possível manter o feixe focado até mesmo na menor dimensão do cristal – uma borda medindo 10 a 20 milionésimos de metro ou menos – enquanto gira.
“Além disso, o detector FMX e outros sistemas operam tão rápido que podemos coletar um conjunto de dados completo em apenas 10 a 15 segundos por amostra, com qualidade suficiente para resolver uma estrutura antes que ocorram danos significativos aos raios X”.
Do congelado ao fisiológico
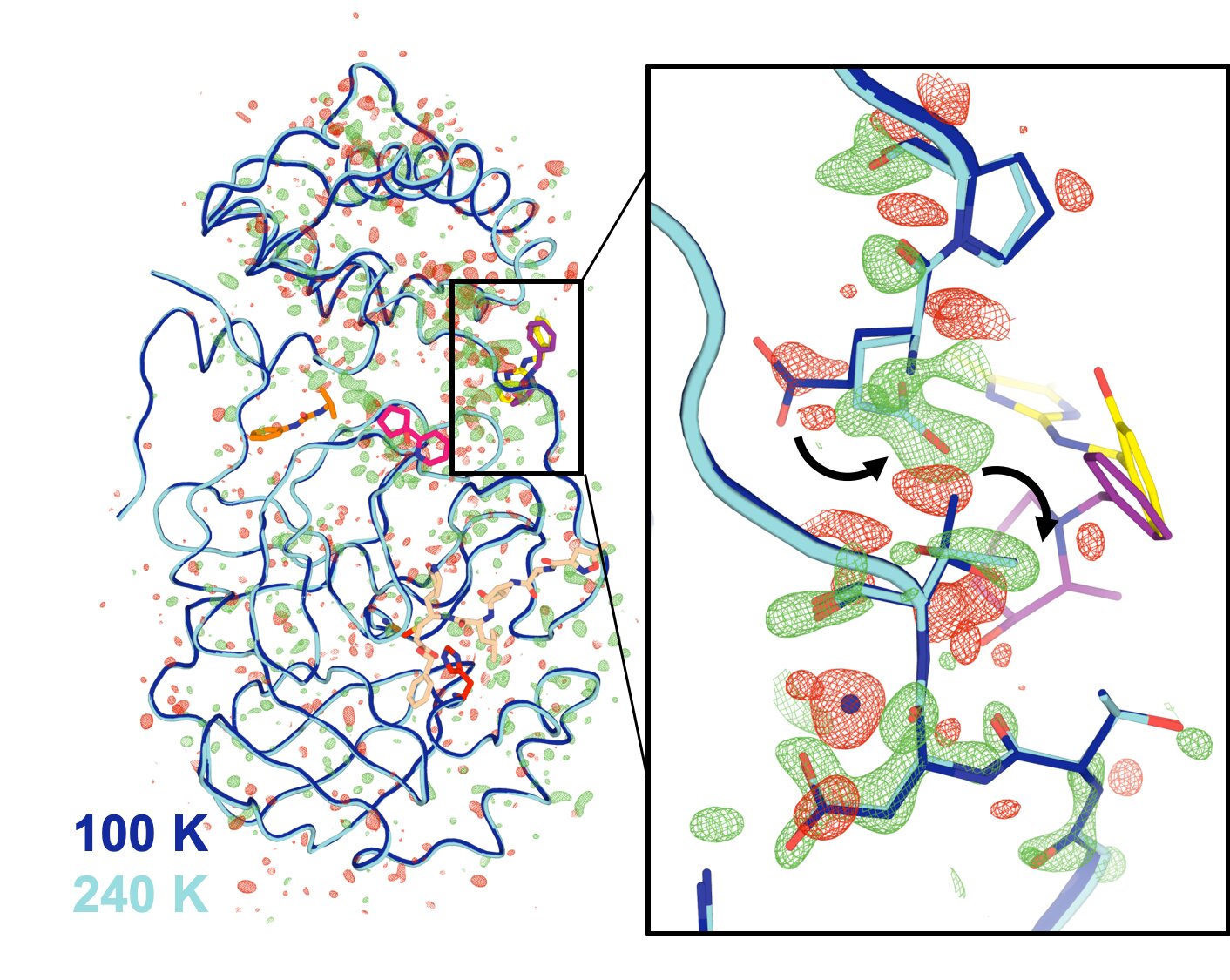
Os cientistas usaram o FMX para coletar a primeira série de dados cristalográficos Mpro em cinco temperaturas diferentes, variando de criogênica (-280 graus Fahrenheit) ao que é frequentemente chamado de “temperatura ambiente” em cristalografia de raios-X (cerca de 39 ° F ) para fisiológico (98°F). Eles também estudaram o cristal à temperatura ambiente sob alta umidade. Em seguida, eles alimentaram os dados em um tipo especial de simulação de computador para identificar muitos arranjos de nível atômico possíveis sob cada conjunto de condições.
Os resultados revelaram mudanças conformacionais sutis, incluindo maior flexibilidade em partes da proteína em temperaturas mais altas. A equipe também viu algumas características que eram exclusivas da enzima sob condições fisiológicas.

A maioria das mudanças não ocorreu diretamente no “sítio ativo” da enzima – a parte que está diretamente envolvida no corte de outras proteínas. Em vez disso, eles estavam em partes da enzima mais distantes desse local. Mas os dados sugerem que esses locais distantes podem afetar o local ativo por meio de uma espécie de mecanismo de controle remoto que é comum em sistemas biológicos, disse Keedy. Desativar até mesmo esses locais distantes poderia bloquear a função da enzima.
“Você pode pensar no Mpro como uma espécie de fita dobrada, composta de duas metades idênticas (formando dímeros) que se unem de maneira simétrica, como um aperto de mão”, disse Keedy. O centro desta região de handshake (a “interface de dímero”) liga-se ao sítio ativo através de uma região de alça flexível da proteína.
Como Keedy explicou, os cientistas descobriram que, em temperaturas mais altas, “o aperto do ‘aperto de mão’ muda – os dois componentes reajustam um pouco o aperto. Isso nos diz que, quando o vírus está nos infectando, pode haver algum tipo de comunicação através deste loop entre a interface do dímero e o site ativo”, disse Keedy.
O caminho para o design de medicamentos
“Vemos mudanças sutis na estrutura neste estudo, mas o design do medicamento depende de mudanças sutis – menos de um bilionésimo de metro aqui, menos de um bilionésimo de metro ali”, observou Keedy.
Outros estudos mostraram que pequenas moléculas semelhantes a drogas podem se ligar à enzima em alguns locais distantes identificados neste novo trabalho.
“Se pudéssemos aperfeiçoar essas moléculas, otimizá-las, elaborá-las, ajustá-las, poderíamos ter um novo ponto de apoio para alterar a função da enzima – não no sítio ativo, pois essencialmente todos os antivirais para essa proteína são atualmente visando, mas em um local diferente por meio de um mecanismo diferente”, disse Keedy. “Nossas descobertas estabeleceram a inspiração para explorar essa ideia”.
O papel da água
Explorar a enzima em umidade elevada também imita as condições fisiológicas dentro das células cheias de água – e pode fornecer pistas adicionais para orientar o projeto de drogas.
“Para esses estudos, depois de selecionar o cristal que queremos estudar, colocamos uma capa especial sobre ele para evitar que seque”, disse Babak Andi, da NSLS-II. “Então, quando colocamos a amostra na linha de luz para coleta de dados de raios-X, removemos a manga e sopramos um fluxo contínuo de ar a 99,5% de umidade sobre o cristal enquanto coletamos os dados”.
Os resultados revelaram moléculas de água específicas que se ligam frouxamente à enzima, incluindo uma próxima ao sítio ativo, apenas sob a condição de alta umidade.
“Essas moléculas de água dão uma dica de onde os inibidores podem se ligar”, disse Andi.
Seu grupo vem estudando uma série de potenciais moléculas semelhantes a drogas que parecem substituir moléculas de água fracamente ligadas quando se ligam perto do sítio ativo do Mpro. Esse trabalho é relatado em um artigo publicado recentemente no Scientific Reports do portfólio de periódicos da Nature.
Ambos os cientistas ficaram gratos pelo espírito colaborativo de toda a equipe, que incluiu cientistas adicionais do Brookhaven Lab e CUNY, bem como da Diamond Light Source no Reino Unido.
“Era muito importante poder ter a colaboração remota para fazer este projeto funcionar, onde tínhamos pessoas no local na linha de luz e pessoas externas que poderiam fazer a modelagem computacional”, disse Keedy. “É um exemplo de muitos durante a pandemia da comunidade científica se unindo”.
Publicado em 18/08/2022 11h25
Artigo original:
Estudo original:


