As vacinações de COVID-19 em grande escala estão em andamento em muitos países em resposta à pandemia de COVID-19. Aqui, relatamos, além da geração de anticorpos neutralizantes, alterações consistentes na hemoglobina A1c, níveis séricos de sódio e potássio, perfis de coagulação e funções renais em voluntários saudáveis após a vacinação com uma vacina SARS-CoV-2 inativada.
Alterações semelhantes também foram relatadas em pacientes com COVID-19, sugerindo que a vacinação mimetizou uma infecção. O sequenciamento de mRNA de célula única (scRNA-seq) de células mononucleares do sangue periférico (PBMCs) antes e 28 dias após a primeira inoculação também revelou alterações consistentes na expressão gênica de muitos tipos diferentes de células imunes. A redução de células T CD8 + e o aumento no conteúdo de monócitos clássicos foram exemplares. Além disso, o scRNA-seq revelou aumento da sinalização de NF-kB e redução das respostas do interferon tipo I, que foram confirmadas por ensaios biológicos e também relatadas como ocorrendo após a infecção por SARS-CoV-2 com sintomas agravantes. Ao todo, nosso estudo recomenda cautela adicional ao vacinar pessoas com condições clínicas pré-existentes, incluindo diabetes, desequilíbrios eletrolíticos, disfunção renal e distúrbios de coagulação.
A pandemia COVID-19 afetou profundamente a humanidade. O desenvolvimento de vacinas COVID-19 em várias formas está em andamento de maneira acelerada e sem precedentes. Apesar de algumas incertezas com relação às consequências potenciais, vacinações em larga escala estão ocorrendo em muitos países. Foram desenvolvidas diferentes vacinas COVID-19, incluindo partículas virais inativadas, vacinas de mRNA, vacinas baseadas em adenovirais e etc. Historicamente, a pesquisa de vacinas tem se concentrado em se a vacinação pode ou não gerar anticorpos neutralizantes para proteger contra infecções virais, enquanto as influências de curto e longo prazo das várias vacinas recentemente desenvolvidas para a fisiopatologia humana e outras perspectivas do sistema imunológico humano não têm foram totalmente investigados.
Com o desenvolvimento da tecnologia de sequenciamento de mRNA de célula única em grande escala (scRNA-seq), a investigação sistemática da função do sistema imunológico das pessoas com precisão tornou-se possível, principalmente por meio de scRNA-seq de células mononucleares do sangue periférico (PBMCs). Durante a pandemia de COVID-19, um grande corpo de estudos usando scRNA-seq de PBMCs revelou mudanças detalhadas na expressão gênica em diferentes subtipos de células imunes, incluindo diferentes tipos de células T e B, células NK, monócitos, células dendríticas, etc. durante e após a infecção, os resultados indicaram uma grande redução do número de células T CD4 + e CD8 + e exaustão de células T após a infecção por SARS-CoV-2. Também foram observados números reduzidos de células T invariáveis associadas à mucosa periférica (MAIT) e sua migração para dentro e para fora do pulmão.
Respostas imunes inflamatórias altamente ativadas, incluindo respostas de interferon-gama (IFN-γ), interleucina-6 (IL-6) e NF-kB, foram relatadas em pacientes com COVID-19 Muitos estudos revelaram diferenças de estado imunológico entre pessoas com sintomas graves e leves, em que as respostas de interferon tipo I forte (IFN-α/β) foram benéficas após a infecção por COVID-19 e as respostas atenuadas de IFN-α/β foram associadas ao desenvolvimento de sintomas graves. Em contraste, respostas inflamatórias de NF-kB mais fortes foram associadas a sintomas mais graves. Além disso, o aumento de células -T e o conteúdo reduzido de neutrófilos foram relatados como associados a sintomas mais leves.
Após infecções por SARS-CoV-2, muitas pessoas desenvolveram vários graus de síndromes respiratórias e algumas com problemas gastrointestinais. Foi relatado que distúrbios de coagulação do sangue, problemas de vasculatura, desequilíbrios eletrolíticos, distúrbios renais, distúrbios metabólicos, etc. eram complicações clínicas importantes com COVID-19. A maneira pela qual a vacinação mimetizaria uma infecção não foi totalmente avaliada.
Neste estudo, recrutamos voluntários saudáveis que deveriam ser vacinados com uma vacina inativada de SARS-CoV-2 (Vero Cell) 3, para participar de testes de anticorpos e anticorpos neutralizantes, bem como medições laboratoriais clínicas detalhadas antes e em momentos diferentes após vacinação (regimes de duas doses com esquemas ligeiramente diferentes foram aplicados).
Para nossa surpresa, observamos mudanças fisiopatológicas bastante consistentes com relação ao conteúdo eletrolítico, perfis de coagulação, função renal, bem como características relacionadas ao metabolismo do colesterol e da glicose, como se essas pessoas tivessem experimentado uma infecção com SARS-CoV-2. Além disso, os resultados de PBMCs scRNA-seq também indicaram reduções consistentes em células T CD8 + e aumentos no conteúdo de monócitos, bem como sinalização inflamatória de NF-kB aprimorada, que também mimetizou respostas após a infecção. Surpreendentemente, as respostas do interferon tipo I, que foram associadas a danos reduzidos após a infecção por SARS-CoV-2 e sintomas mais leves, pareceram ser reduzidas após a vacinação, pelo menos 28 dias após a primeira inoculação. Isso pode sugerir que no curto prazo (1 mês) após a vacinação, o sistema imunológico de uma pessoa está em um estado não privilegiado e pode exigir mais proteção.
Resultados
Acompanhamento longitudinal da produção de anticorpos anti-SARS-CoV-2 e anticorpos neutralizantes após a inoculação da vacina SARS-CoV-2 inativada
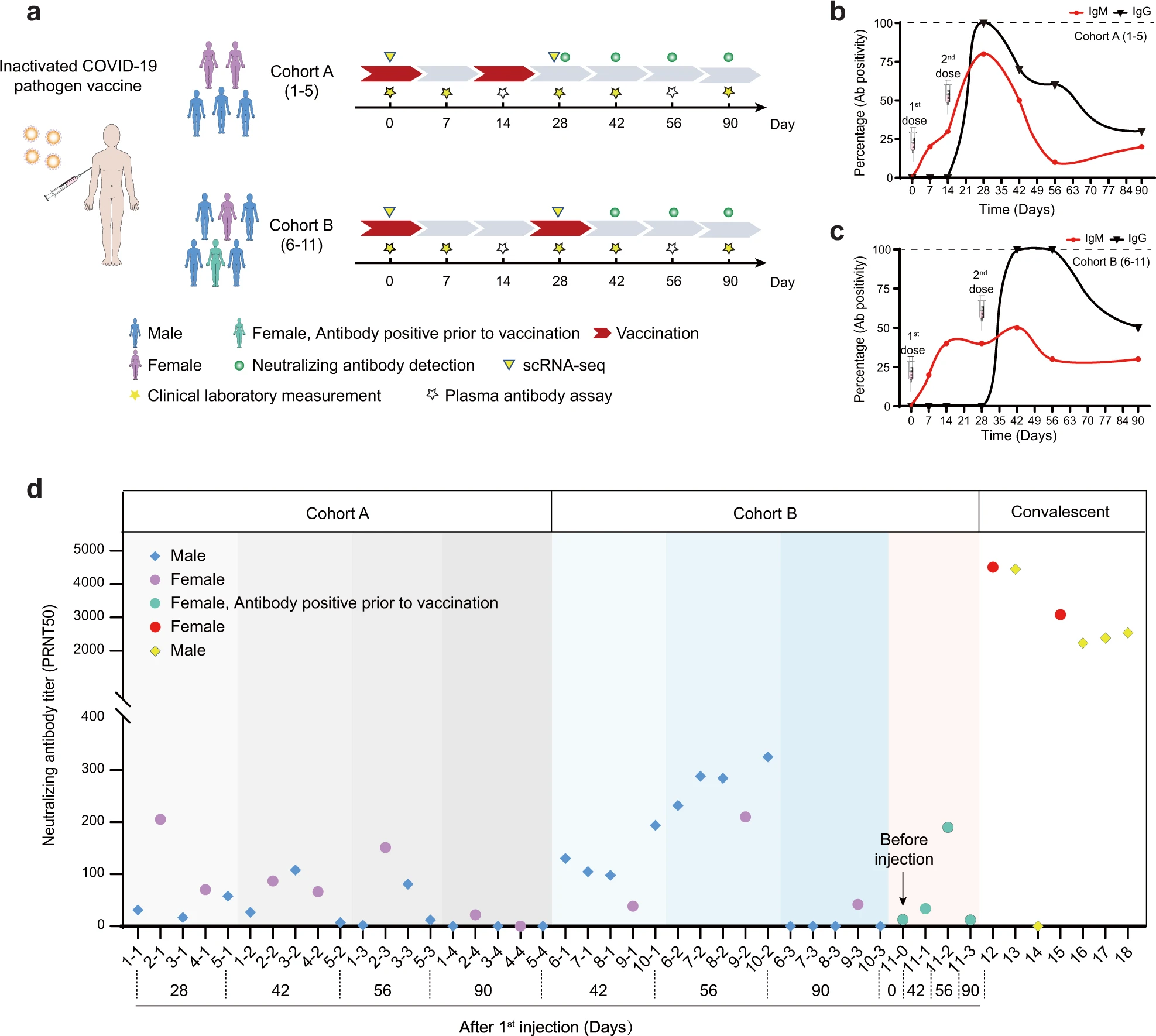
Um total de 11 voluntários adultos saudáveis de ambos os sexos, com idades entre 24-47 anos, com um IMC de 21,5-30,0 kg / m², foram incluídos neste estudo (Fig. 1a e Tabelas Suplementares S1 e S2). A vacina SARS-CoV-2 (Vero Cell), inativada (Beijing Institute of Biological Products Co. Ltd), foi administrada por via intramuscular no deltóide. Os voluntários foram divididos em duas coortes; cinco participantes (coorte A) foram vacinados com uma dose completa (4 µg) de vacina inativada SARS-CoV-2 (Vero Cell) nos dias 1 e 14, e seis participantes (coorte B) receberam uma dose completa da vacina nos dias 1 e 28 (Fig. 1a). Um dos voluntários do grupo B foi testado positivo para anti-SARS-CoV-2 IgM e IgG logo antes da vacinação, o que sugere possíveis infecções anteriores. No entanto, não houve registro de positividade anterior por diagnóstico de ácido nucleico (NA) para COVID-19 (marcado em verde na Fig. 1a). Para todos os exames de acompanhamento, os dados desse indivíduo foram marcados em verde para rastrear quaisquer possíveis influências de infecções anteriores em potencial.
Os eventos adversos foram monitorados diariamente durante os primeiros 7 dias após cada inoculação e, em seguida, registrados pelos participantes em cartões de diário nas semanas seguintes. No geral, as reações adversas foram leves (graus 1 ou 2) e transitórias (Tabela Suplementar S3). As amostras de sangue foram coletadas nos dias 0, 7, 14, 28, 42, 56 e 90, e as amostras de urina foram coletadas nos dias 0, 14, 28, 42 e 90. As amostras de plasma foram submetidas a anti-SARS-CoV- 2 Teste de IgM / IgG usando vários kits de diagnóstico, os resultados do kit mais sensível foram usados para quantificação (Fig. 1b, c). Os resultados do teste da coorte A demonstraram que antes da 2ª inoculação 0% dos participantes desenvolveram anti-SARS-CoV-2 IgG, mas no dia 28, que foi 2 semanas após a 2ª inoculação, 100% dos participantes tiveram teste positivo ( Fig. 1b).
No geral, o IgM apareceu mais cedo do que o IgG, o que era esperado. A positividade de IgG e IgM diminuiu no dia 42 e permaneceu em níveis relativamente baixos no dia 90 na coorte A. Para a coorte B, ninguém desenvolveu IgG até após a 2ª inoculação. No entanto, no dia 42, a positividade de IgG atingiu 100% (Fig. 1c) e se manteve até o dia 56, sugerindo que o protocolo de vacinação para a coorte B foi mais eficaz. No dia 90, a positividade de IgG também reduziu para 50%, indicando que a produção de anticorpos não se sustentou por um longo tempo. Além disso, realizamos testes para anticorpos neutralizantes de SARS-CoV-2 (Fig. 1d), e os resultados também indicaram que duas inoculações com 28 dias de intervalo (coorte B) resultaram em títulos de anticorpos protetores mais elevados em comparação com duas inoculações com 14 dias de intervalo (coorte UMA). Por outro lado, parecia que os títulos de anticorpos neutralizantes anti-SARS-CoV-2 eram geralmente mais baixos do que aqueles em indivíduos convalescentes COVID-19, conforme relatado antes3 (Fig. 1d). Por 90 dias, os títulos de anticorpos neutralizantes diminuíram dramaticamente em todos os voluntários (Fig. 1d). Curiosamente, o indivíduo que era positivo para anticorpos antes da vacinação não era mais propenso a gerar anticorpos neutralizantes em comparação com o resto dos participantes, sugerindo que a infecção potencial anterior pode não ter ocorrido ou pode não gerar proteção duradoura na perspectiva de neutralização produção de anticorpos.
Alterações nas medições laboratoriais clínicas após a vacinação
Testes de rotina de laboratório clínico, incluindo índices relacionados à infecção, parâmetros hematológicos, função de coagulação, glicose no sangue, lipídios séricos, enzimas relacionadas à função cardíaca, eletrólitos, fígado e biomarcadores relacionados à função renal, foram medidos para revelar as características de segurança da vacina (Fig. . 2a e Tabelas Suplementares S4 e S5). A contagem de leucócitos aumentou significativamente, embora apenas ligeiramente, após a vacinação no dia 7. Não foram detectadas diferenças nos pontos de tempo seguintes (Fig. 2b). Para nossa surpresa, aumentos bastante consistentes nos níveis de HbA1c foram observados em voluntários saudáveis, independentemente de pertencerem à coorte A ou B. No dia 28 após a primeira inoculação, três de 11 indivíduos atingiram a faixa pré-diabética (Fig. 2c). Nos dias 42 e 90, os níveis médios de HbA1c pareceram reverter, mas ainda eram significativamente mais elevados do que antes da vacinação. Trabalhos anteriores demonstraram que pacientes diabéticos com níveis de glicose no sangue não controlados são mais propensos a desenvolver formas graves de COVID-19. Foi demonstrado que níveis elevados de glicose no sangue / glicólise promovem a replicação do SARS-CoV-2 em monócitos humanos por meio da produção de espécies reativas de oxigênio mitocondrial e ativação de HIF1A, apresentando, portanto, uma característica desvantajosa.
Fig. 2: Mudanças temporais das medidas laboratoriais clínicas após a vacinação.

Os níveis de potássio sérico diminuíram significativamente nos dias 28, 4 e 90 após a primeira inoculação, com uma amostra abaixo do limite normal inferior no dia 42 (Fig. 2d, painel esquerdo). Da mesma forma, os níveis séricos de sódio também diminuíram após a vacinação (Fig. 2d, painel direito), indicativo das influências da vacina no equilíbrio eletrolítico. Novamente, o desequilíbrio eletrolítico também foi associado ao COVID-1921. Coagulopatia é outra condição clínica induzida por COVID-19. Descobrimos que os perfis de coagulação mudaram significativamente após a vacinação, no curto prazo (7 dias) após a 1ª inoculação, os perfis de coagulação tendiam a diminuir o Tempo de Protrombina (PT), enquanto o efeito a longo prazo (28 e 42 dias) era próximo tempo de tromboplastina parcial ativada (APTT) e prolongamento do TP (Fig. 2e). No dia 90, os perfis voltaram aos anteriores à vacinação (Fig. 2e). Além disso, encontramos níveis elevados de colesterol no sangue nos dias 7, 28 após a primeira inoculação, e níveis elevados de ácido biliar total também foram detectados no dia 7 (Fig. 2f, g). A disfunção renal é outra condição clínica associada ao COVID-19 e, em 28, 42 e 90 dias após a primeira inoculação, os níveis de creatinina sérica estavam significativamente mais elevados do que antes da vacinação, resultando em eTFG reduzida (Fig. 2h). Foi relatado que a maioria dessas características clínicas está associada ao desenvolvimento de sintomas graves em pacientes com COVID-19 (Tabela Suplementar S6). No geral, não houve diferenças estatisticamente significativas entre as coortes A e B, exceto para apenas alguns índices (Tabela Suplementar S7), portanto, os dados de duas coortes foram agrupados para apresentação de dados clínicos e análises subsequentes.
O scRNA-seq revelou alterações dramáticas na expressão gênica de quase todas as células do sistema imunológico após a vacinação
Para explorar as características imunológicas de voluntários saudáveis após a vacinação, realizamos scRNA-seq baseado em gotículas (10 × Genomics) para estudar perfis transcriptômicos de PBMCs de voluntários pertencentes à coorte A ou B, antes e 28 dias após a vacinação (Fig. 3a e Suplementar Fig. S1a). Após o pré-processamento e eliminação de células de baixa qualidade (consulte “Materiais e métodos”), obtivemos 188.886 células de todas as amostras de PBMC, entre as quais 86.685 células eram da coorte A e 102.201 células da coorte B. Todas as células qualificadas foram integradas ao conjunto de dados unificado e submetidos a análises posteriores.
Fig. 3: Alterações no tipo de célula imunológica periférica e composições de subtipo, bem como na expressão gênica antes e 28 dias após a primeira inoculação.

Usando agrupamento baseado em gráfico de aproximação e projeção de variedade uniforme (UMAP), algoritmo de reconhecimento de célula única de tipos de células (SingleR) e anotação manual com base em marcadores de genes canônicos, identificamos 22 tipos de células ou subtipos e realizamos análise de expressão diferencial entre todos os tipos de células (Fig. 3b e Tabela Suplementar S8). As células (transcriptomas celulares) das amostras antes (azul) e depois da vacinação (laranja) foram distintamente separadas na representação UMAP para ambas as coortes, o que significa que as características imunológicas mudaram drasticamente em quase todos os tipos de células imunológicas detectados e de forma consistente em todos os voluntários ( Fig. 3c).
Entre os 11 pares (antes e depois) de amostras de PBMC, 10 pares foram sequenciados juntos e um par foi sequenciado separadamente em um lote diferente. As distribuições de UMAP foram drasticamente semelhantes, independentemente dos diferentes lotes, sugerindo efeitos de lote de sequenciamento mínimo (Fig. Suplementar S1b). Dois lotes independentes de sequenciamento revelaram mudanças semelhantes antes e depois da vacinação, sugerindo que as mudanças são reais, enquanto o uso do método de correção de efeito de lote (Harmony) (Fig. Suplementar S1c-e) resultaria em excesso de filtração e eliminação das mudanças reais causadas por vacinação. Além disso, o agrupamento da amostra com base no coeficiente de correlação de Pearson dos transcriptomas indicou que as amostras das duas coortes (A e B) se misturaram bem antes e depois da vacinação, enquanto as alterações induzidas pela vacinação puderam ser claramente observadas (Fig. 3d) . Portanto, para aumentar o poder estatístico, combinamos as duas coortes para análises subsequentes.
Para revelar as diferenças nas composições do tipo de célula antes e após a vacinação, calculamos as porcentagens relativas de todos os tipos de células em PBMCs de cada indivíduo com base nos dados de scRNA-seq (Fig. 3e). Observamos diminuições no conteúdo de células T reguladoras CD4 + (CD4.Treg), células T CD8 + (CD8.T) e células CD8 + em proliferação (CD8.Tprolif) após a vacinação (Fig. 3e). Diminuições nos conteúdos de células -T (gd.T.Vd2) também foram significativas (Fig. 3e). Em contraste, a vacinação aumentou o conteúdo de monócitos clássicos CD14 + (Mono.C) (Fig. 3e), consistente com as medições de laboratório clínico (Fig. 3f). O conteúdo geral de linfócitos, que incluiu todas as células T CD4 +, todas as células T CD8 +, células B e células NK, não mudou significativamente antes e após a vacinação, o que também foi confirmado por medições laboratoriais clínicas (Fig. 3g). Coletamos um conjunto de dados publicado de 196 pacientes infectados com COVID-19 e controles7 e analisamos nossos dados junto com esse conjunto de dados. O resultado indicou que as mudanças induzidas pela vacinação no conteúdo celular de todos os cinco subtipos de células imunes diferentes também mudaram nas mesmas direções em pacientes COVID-19 em comparação com os controles, exceto para células T CD8 + em proliferação (Fig. Suplementar S2).
Para estudar as alterações detalhadas da expressão gênica induzida pela vacinação, combinamos amostras individuais em amostras pseudo-bulk e usamos o teste de amostra pareada para identificar genes diferencialmente expressos (DEGs) (Fig. 3h e Tabela Suplementar S9). Genes significativamente regulados positivamente estavam envolvidos na “sinalização de TNFα via NF-kB”, “respostas inflamatórias” e “interação citocina-receptor de citocina”, “sinalização IL6-JAK STAT3”, “coagulação”, “hipóxia”, que foi relatada por COVID-19, enquanto as vias relacionadas ao ciclo celular foram reguladas para baixo (Fig. 3i). Esses resultados apoiaram a noção de que a vacinação mimetizou uma infecção
As alterações de expressão gênica específicas do subtipo de célula imune em destaque refletem as alterações de laboratório clínico
Antes da elucidação da heterogeneidade funcional e alterações de expressão gênica específica do tipo de célula entre as amostras antes e depois da vacinação, agrupamos as células em 11 tipos principais: (1) células T CD4 + de estado ingênuo, (2) T CD8 + T de estado ingênuo células, (3) células T auxiliares CD4 + (incluindo CD4.T, CD4.Treg e CD4.Tprolif), (4) células T citotóxicas CD8 + (incluindo CD8.T, CD8B.T e CD8.Tprolif), (5 ) MAIT, (6) células -T, (7) células NK (incluindo NK, NK proliferativa), (8) células B / plasmablast (incluindo células B e plasmablastos), (9) monócitos / células dendríticas (incluindo mono clássicas , mono intermediário, mono não clássico, DC1 mieloide, DC2 mieloide e DC plasmocitoide), (10) células T efetoras terminais CD4 + e (11) células T efetoras terminais CD8 +. Após onze principais categorizações de tipo de célula, realizamos comparações em nível de amostra agregando a expressão gênica entre os principais tipos de células em cada doador e, em seguida, realizamos uma análise de expressão diferencial usando muscat. Identificamos genes expressos diferencialmente (DEGs) entre todos os principais tipos de células (Fig. 4a e Tabela Suplementar S10) e conduzimos a análise funcional do gene (Fig. 4b).
Ecoando os resultados da medição clínica, genes relacionados a “homeostase do colesterol”, “coagulação” e “resposta inflamatória” (CXCL8, CD14, IL6 e TNFRSF1B), “sinalização de TNFα via NF-kB” (NFKB1, NFKB2, NFKBIE, TNFAIP3 e TNFSF9) e “hipóxia” (HIF1A) foram regulados positivamente. Além disso, genes relacionados à “sinalização de TGFβ”, “sinalização IL2-STAT5” (IFNGR1, MAPKAPK2 e CASP3) e “sinalização IL6-JAK-STAT3” também foram regulados positivamente (Fig. 4c). Para visualizar quais tipos de células foram enriquecidos para essas assinaturas, realizamos a pontuação do módulo do gene e exibimos as pontuações nas coordenadas UMAP, bem como gráficos de caixa agrupados (Fig. 4c e Tabela Suplementar S11). Curiosamente, os genes de “resposta inflamatória” foram altamente expressos em monócitos e após a vacinação aumentaram ainda mais (Fig. 4c), sugerindo que os monócitos eram um dos principais tipos de células que participam das respostas inflamatórias após a vacinação. Em contraste, os genes relacionados à “glicólise”, “metabolismo do ácido biliar” e “resposta do interferon tipo I (IFN-α/β)” foram regulados para baixo, de acordo com nossos dados clínicos e a fisiopatologia de COVID-1913 (Fig. 4d) .
Fig. 4: Análises de expressão diferencial de genes específicos de subtipo e de sobre-representação de conjuntos de genes que descrevem mudanças comuns de expressão de genes entre diferentes tipos de células imunes após a vacinação.

As alterações mais comuns em vários subtipos de células imunes revelaram aumentos na sinalização de NF-kB e diminuições nas respostas de IFN-α/β Dado que os grupos de genes mudaram sua expressão dramaticamente entre todos os principais tipos de células, formulamos a hipótese de que pode haver alguns fatores de transcrição atuando como reguladores principais que levam a alterações imunológicas. Para resolver os desafios computacionais associados a um grande conjunto de dados, usamos o algoritmo MetaCell para agregar grupos homogêneos de células em metacélulas e, finalmente, produzimos 1857 metacélulas (893 antes e 964 após a vacinação) para representar toda a estrutura dos dados de scRNA-seq (Fig. 5a). Essas metacélulas foram então aplicadas à “inferência e agrupamento de redes regulatórias de célula única (SCENIC)” para construir as redes regulatórias de genes. O fluxo de trabalho produziu uma lista de 157 “regulons”, que incluía fatores de transcrição e seus alvos diretos. As atividades do regulon foram pontuadas usando AUCell para acessar o enriquecimento médio de todos os genes pertencentes a cada regulon em cada metacel, bem como o enriquecimento do gene regulon médio em todas as 893 metacélulas antes da vacinação e 964 metacélulas após a vacinação. Foram identificados oito regulons regulados positivamente e oito regulons regulados negativamente no topo da classificação (mais ativos) após a vacinação (Fig. 5b). Selecionamos 3 + 3 regulons típicos para construir uma rede regulatória, conforme apresentado na Fig. 5c (Tabela Suplementar S12). A rede mostrou dois grupos distintos, um é composto por IRF2, STAT1 e STAT2, que foram regulados para baixo após a vacinação, e o outro, continha RELB, NFKB2 e HIF1A, que foram regulados positivamente após a inoculação. Os termos GO da rede regulada positivamente estão predominantemente relacionados à diferenciação, ativação e “Formação do centro germinativo” de linfócitos, o que sugere que as células T e B foram ativadas após a vacinação. Além disso, a sinalização de NF-kB também foi elevada após a vacinação. A rede regulada para baixo foi enriquecida para muitas vias relacionadas aos interferons e secreção de citocinas (Fig. 5d e Tabela Suplementar S13). Isso sugeriu que a vacinação pode inibir as respostas do interferon no sistema imunológico periférico, reduzindo as atividades dos regulons STAT1, STAT2 e IRF2, que se pensava serem fatores mestres de transcrição que direcionam a sinalização do interferon tipo I e III.
Fig. 5: Identificação dos regulons principais e suas redes regulatórias antes e depois da vacinação.

Para confirmar a inibição induzida por vacinação de respostas de interferon reveladas por scRNA-seq, nós estimulamos PBMCs de indivíduos vacinados antes e 28 dias após a vacinação com IFN-α/β. Após 16 h de cultura e 12 h de estimulação, usamos RT-qPCR para medir a expressão relativa dos reguladores principais IRF2, IRF7 e STAT2. STAT2 e IRF7 foram significativamente regulados para baixo após a vacinação, mas IRF2 mostrou uma tendência de regulação para baixo (Fig. 5e, f). As análises do regulon indicaram que os estados do sistema imunológico periférico após a vacinação reduziram as respostas do interferon tipo I, indicativo de habilidades antivirais gerais atenuadas pelo menos 28 dias após a primeira inoculação.
Respostas inflamatórias induzidas por vacinação em monócitos
Relatórios recentes descreveram assinaturas conservadas de resposta imune do hospedeiro a infecções respiratórias virais, a saber, a assinatura de meta-vírus (MVS), que também é conservada na infecção por SARS-CoV-2. Pontuações mais altas de MVS estão associadas à infecção. Ao todo, 380 (158 positivamente e 222 contribuíram negativamente para as pontuações de MVS) de 396 (161 positivamente e 235 contribuíram negativamente) genes selecionados para medição de MVS foram detectados em nosso conjunto de dados. Para investigar as respostas imunes do hospedeiro após a vacinação com SARS-CoV-2 inativado, separamos os conjuntos de genes positivos e negativos e calculamos as pontuações MVS (Fig. 6a). As pontuações de MVS foram substancialmente mais altas após a vacinação (Fig. 6b, c), sugerindo que a vacinação simulou uma infecção. Curiosamente, o conjunto de genes MVS positivo foi predominantemente expresso em monócitos, enquanto o conjunto negativo em linfócitos, indicando que diferentes respostas imunes específicas do tipo de célula ocorreriam após a vacinação (Suplementar Fig. S3a, b).
Fig. 6: Os monócitos exibiram pontuações de MVS altas e vias correlacionadas com pontuação de MVS.

Para investigar quais vias foram associadas com o conjunto de genes MVS-positivo e o conjunto de genes MVS-negativo, calculamos a correlação de Spearman entre as pontuações dos conjuntos de genes MVS e as vias diferencialmente enriquecidas previamente identificadas usando nossos dados de scRNA-seq (Fig. 6d). A via mais altamente correlacionada com a pontuação MVS e conjunto positivo para MVS foi “Sinalização de resposta inflamatória”, que foi significativamente regulada positivamente em monócitos após a vacinação, juntamente com CD14, FPR1, C5AR1, NAMPT, NLRP3, CDKN1A e IFNGR2. Considerando que o conjunto negativo de MVS correlacionou-se bem com a “assinatura de citotoxicidade”, representada por NKG7, CCL4, CST7, PRF1, GZMA, GZMB, IFNG e expressão de CCL3, diminuiu significativamente em muitos subtipos de células T, mas não em células NK após a vacinação (Suplementar Fig. S3c).
Discussão
Esta é uma investigação abrangente das alterações fisiopatológicas, incluindo alterações imunológicas detalhadas em pessoas após a vacinação com COVID-19. Os resultados indicaram que a vacinação, além de estimular a geração de anticorpos neutralizantes, também influenciou vários indicadores de saúde, incluindo aqueles relacionados a diabetes, disfunção renal, metabolismo do colesterol, problemas de coagulação, desequilíbrio eletrolítico, de forma como se os voluntários tivessem uma infecção. O scRNA-seq de PBMCs de voluntários antes e depois da vacinação revelou mudanças dramáticas na expressão do gene da célula imune, não apenas ecoando algumas das medidas laboratoriais clínicas, mas também sugestivo de aumento das respostas inflamatórias relacionadas ao NF-kB, que acabou ocorrendo principalmente em monócitos clássicos. A vacinação também aumentou o conteúdo clássico de monócitos. Além disso, o conjunto de genes contribuindo positivamente para os escores de MVS, também conhecido por estar associado ao desenvolvimento de sintomas graves, foi altamente expresso em monócitos. As respostas do interferon tipo I (IFN-α/β), supostamente benéficas contra COVID-19, foram reguladas negativamente após a vacinação. Além disso, os genes MVS negativos foram altamente expressos em linfócitos (células T, B e NK), mas apresentaram expressão reduzida após a vacinação. Juntos, esses dados sugeriram que após a vacinação, pelo menos no dia 28, além da geração de anticorpos neutralizantes, o sistema imunológico das pessoas, incluindo os de linfócitos e monócitos, talvez estivesse em um estado mais vulnerável.
Curiosamente, nossos dados preliminares demonstraram que se pré-incubássemos RBD de SARS-CoV-2 com os PBMCs (de voluntários antes e depois da vacinação) e, em seguida, tratássemos as células com IFN-α/β, as respostas do interferon tipo I seriam realmente aumentadas em PBMCs após a vacinação, sugerindo que talvez a vacinação, embora reduza a capacidade antiviral geral de uma pessoa, aumenta a função imune adaptativa especificamente para SARS-CoV-2 (Fig. Suplementar S4a). Por outro lado, comparando PBMCs antes da vacinação, o pré-tratamento de SARS-CoV-2 S-RBD pareceu reduzir as respostas do interferon tipo I (P <0,05, IRF2, IRF7, STAT2) (Fig. Suplementar S4b), sugerindo 1ª vez a exposição do peptídeo viral realmente causaria uma redução nas respostas do interferon tipo I em PBMC. Esses dados in vitro suportaram bem os resultados de scRNA-seq.
Vale ressaltar que um indivíduo da coorte A que estava em uso de antibióticos não apresentou redução da expressão gênica associada às respostas do interferon tipo I, e esse indivíduo também apresentou o maior título de anticorpos neutralizantes da coorte. Além disso, calculamos o coeficiente de correlação de Pearson entre os títulos de anticorpos neutralizantes e as respostas inflamatórias medidas pela média da expressão gênica de genes associados à sinalização de TNFα via NF-kB e respostas de interferon-α (interferon tipo I). Os resultados foram 0,32 e 0,39 com P> 0,05 (Fig. Suplementar S4c), respectivamente, sugerindo que as alterações da resposta imune e a proteção imune adaptativa da vacina não parecem estar altamente correlacionadas. Ainda não foi determinado se os antibióticos podem influenciar a eficácia da vacina. Também é bastante interessante que, embora as coortes A e B tivessem diferentes perfis de produção de anticorpos anti-SARS-CoV-2, seus resultados de scRNA-seq de PBMCs foram drasticamente semelhantes, incluindo seus dados de scRNA-seq de células B (Fig. Suplementar S5a-c ) Deve-se notar que após a vacinação, a maioria das células B responsivas, particularmente aquelas que produzem anticorpos anti-COVID-19 maduros (IgG), incluindo células B de memória, devem estar localizadas principalmente em tecidos linfáticos periféricos, como nódulos linfáticos e baço, enquanto apenas algumas células B maduras existiriam na circulação. Portanto, a população de células B em preparações de PBMCs pode não refletir todo o espectro da imunidade humoral.
As análises apresentadas neste estudo, particularmente, scRNA-seq de PBMCs não foram realizadas para avaliações de vacinas anteriores, se as alterações nos genes relacionados à função do sistema imunológico eram específicas de COVID-19 ou poderiam ser geralmente aplicadas a outras vacinas ou outros tipos de vacinas COVID-19 ainda não foram determinadas. No entanto, esses tipos de análises detalhadas devem ser benéficos para o desenvolvimento e aplicações de vacinas. Nosso estudo postula que é imperativo considerar o impacto potencial de longo prazo da vacinação em certas condições médicas ou na saúde humana em geral.
Materiais e métodos
Participantes, coleta de dados clínicos e procedimentos
Voluntários adultos saudáveis foram recrutados para o programa. Todos os indivíduos foram submetidos a um exame físico e preencheram um questionário por médicos treinados. Um adulto saudável de 18 a 60 anos, com temperatura axilar ≤ 37,0 ° C, negativo para o teste de ácido nucleico de SARS-CoV-2 e disposto a concluir todos os processos de estudo programados foram incluídos no estudo. Pessoas com epilepsia, doenças cerebrais ou mentais, história de alergias, doenças crônicas importantes não controladas e achados anormais clinicamente significativos na bioquímica, testes de hematologia foram excluídos. Mulheres grávidas ou amamentando também foram excluídas. Este estudo foi aprovado pelo Comitê de Ética do Shanghai East Hospital de acordo com os princípios da Declaração de Helsinque (nº 2020 (096)). Consentimentos informados por escrito foram obtidos de todos os participantes antes da inscrição.
Um total de 11 participantes foram inscritos e vacinados para avaliar a segurança clínica e as mudanças dinâmicas no sistema imunológico. Entre estes, cinco participantes (coorte A) foram vacinados com 4 μg de dose de vacina inativada SARS-CoV-2 (Vero Cell) nos dias 1 e 14, e seis participantes (coorte B) receberam uma dose de 4 μg da vacina nos dias 1 e 28. Vacina SARS-CoV-2 inativada (Vero Cell) (China Biotechnology Group Corporation) foi administrada por via intramuscular no deltóide. Todas as vacinas foram aprovadas pelos Institutos Nacionais de Controle de Alimentos e Medicamentos da China.
Testes de segurança laboratorial, incluindo índices relacionados à infecção (proteína C reativa, proteína amilóide A sérica), parâmetros hematológicos (contagem de leucócitos, contagem de neutrófilos, contagem de linfócitos, contagem de monócitos, contagem de glóbulos vermelhos, hemoglobina, contagem de plaquetas), função de coagulação índices relacionados (tempo de protrombina, tempo de tromboplastina parcial ativada / APTT, fibrinogênio, atividade de protrombina / PT, razão normalizada internacional / INR), parâmetros relacionados à glicose no sangue (glicose plasmática em jejum, HbA1c), lipídio sérico (colesterol total, triglicerídeo, HDL -C, LDL-C), enzimas relacionadas à função cardíaca (creatinina quinase, CK-MB), eletrólitos (potássio, sódio, cloreto, bicarbonato, cálcio total, magnésio), biomarcadores relacionados à função hepática (por exemplo, albumina, alanina aminotransferase / ALT, aspartato aminotransferase / AST, bilirrubina total e etc.), marcadores relacionados à função renal (creatinina, ácido úrico, nitrogênio urinário / BUN, taxa de filtração glomerular estimada / eGFR) foram medidos.
Teste de anticorpos neutralizantes por PRNT
Cada amostra de soro foi testada usando um ensaio de teste de neutralização por redução de placa (PRNT) para SARS-CoV-2 (2019-nCoV-WIV04) no laboratório BSL-3. Resumidamente, os soros foram inativados por calor a 56 ° C por 30 min e diluídos a 1:50, seguido por diluições em série de três vezes (1:50, 1: 150, 1: 450, 1: 1350, 1: 4050 e 1: 12.150). Os soros foram então misturados com 100 PFU de vírus e incubados a 37 ° C durante 1 h. As misturas de diluição de vírus-soro e o controle de vírus foram então inoculados em monocamadas de células Vero E6 em placas de 24 poços por 1 h antes de adicionar um meio de cobertura incluindo 1,5% de metilcelulose a 37 ° C por 4-5 dias para permitir o desenvolvimento da placa. Em seguida, as placas foram fixadas e coradas com 2% de violeta de cristal em 30% de metanol por 30 min em temperatura ambiente, e as placas foram contadas e medidas manualmente. O título de PRNT foi calculado com base em uma redução de 50% na contagem de placas (PRNT50).
Preparação de suspensões de célula única, preparação de biblioteca de RNA de célula única e sequenciamento
Os PBMCs foram isolados de sangue venoso heparinizado de voluntários saudáveis usando um meio Ficoll-PaqueTM PLUS (GE Healthcare Inc.) de acordo com o método de centrifugação de gradiente de densidade padrão fornecido pelo fabricante. PBMCs foram congelados em meio de congelamento (70% RPMI-1640, 20% FBS e 10% DMSO) e armazenados em nitrogênio líquido até o uso. A captura de uma única célula e a construção da biblioteca foram realizadas usando o kit Chromium Single Cell 5´ Library & Gel Bead (10 × Genomics) de acordo com as instruções do fabricante. As bibliotecas foram sequenciadas usando a plataforma Novaseq 6000 (Illumina).
Análise e estatística de dados scRNA-seq
Dados de sequenciamento de célula única foram alinhados e quantificados usando kallisto / bustools (KB, v0.25.0) 35 contra o genoma de referência humano GRCh38 baixado do site oficial 10 × Genomics. As contagens preliminares foram então usadas para análises a jusante. Fizemos um pipeline para processar os dados. Resumidamente, células com menos de 200 genes foram filtradas, as contagens logarítmicas normalizadas e a seleção dos 3000 genes altamente variáveis (HVGs) principais foram realizadas por Scanpy.
Excluímos genes específicos de HVGs, incluindo genes mitocondriais, genes de imunoglobulina e genes ligados a modelos de transcrição com suporte insuficiente (anotados com o prefixo “Rp-“). Em seguida, a análise de componentes principais (PCA) foi realizada utilizando os HVGs e o algoritmo Harmony foi usado para remover os efeitos do lote25. Usamos a abordagem PARC para identificar clusters e recursos selecionados pela função “FeatureSelectionByEnrichment” do algoritmo cytograph2, seguido por outra rodada de PCA, Harmony e PARC. Posteriormente, calculamos K vizinhos mais próximos em um gráfico KNN, realizamos aproximação e projeção de variedade uniforme (UMAP) por Pegasuspy e identificamos clusters por PARC. Além disso, aplicamos Scrublet para identificar dupletos potenciais.
O controle de qualidade foi aplicado aos clusters com base na saída da primeira rodada do pipeline:
1. Aglomerados com mais de 20% de células, com pontuação de dupleto> 0,4, foram definidos como grupos de dupletos.
2. Clusters com mais de 20% de células que tinham> 20% de seus transcritos mapeados para genes mitocondriais foram definidos como clusters de baixa qualidade.
3. Clusters com mais de 20% de células que tiveram <0,05% de seus transcritos mapeados para genes mitocondriais foram definidos como núcleos.
4. Expressão mediana de PPBP, PF4, HBB, HBA2> 0, indicando eritrócitos e plaquetas.
5. Menos de 50 células.
6. Números de genes detectados <1000.
7. Razão da média do total de UMIs e média dos genes detectados <2.
8. Scrublet identificou dupletos.
9. Usando DBSCAN para remover outliers.
Depois de remover as células de baixa qualidade, anotamos as células pelo algoritmo de reconhecimento de tipos de células (SingleR), referindo-se aos conjuntos de dados imunes do Monaco.
As células qualificadas foram submetidas a análise a jusante. Da mesma forma, executamos novamente o pipeline para identificar os principais tipos de células, incluindo células T (CD3D, CD3E, CD3G, CD40LG, CD8A, CD8B), células B (MS4A1, CD79A, CD79B), células NK (GNLY, NKG7, TYROBP, NCAM1), e monócitos (CST3, LYZ). Além disso, executamos o pipeline em cada tipo de células, respectivamente, e ainda identificamos subtipos com base nos tipos de células identificados com SingleR e marcadores bem caracterizados (Fig. 3b).
Comparando a proporção de células imunes
Para amostras de PBMCs, calculamos as proporções das células imunes para cada tipo de célula principal e subtipos subjacentes. Para cada amostra, a proporção do tipo de célula foi calculada pelo número de células em um determinado tipo de célula dividido pelo número total de células. Para identificar mudanças nas proporções celulares entre amostras em grupos diferentes, realizamos um teste de Wilcoxon nas proporções de cada um dos principais tipos de células, bem como nos subtipos de células em diferentes grupos (Fig. S2 suplementar). Apenas os tipos de células com diferenças estatisticamente significativas (P <0,05) em proporções são mostrados na Fig. 3e.
Análise de expressão diferencial, análise de super-representação de conjuntos de genes e módulos de assinatura de pontuação
Para investigar alterações de características imunológicas, identificamos DEGs pelo algoritmo muscat26 com parâmetros padrão. Resumidamente, primeiro colapsamos a soma dos dados, somando UMIs entre células para cada doador saudável, para produzir um perfil de UMIs de estilo seq de RNA em massa para cada amostra. Posteriormente, as contagens agregadas foram carregadas na função pbDS para identificar DEGs, e os mapas de calor foram plotados pela função pbHeatmap. A análise de super-representação do conjunto de genes de DEGs (logFC> 0,5 e P <0,05 ajustado) foi realizada usando o teste exato de Fisher unilateral (conforme implementado no pacote "gsfisher" R) com o gene "HALLMARK", "KEGG" e "REACTOME" conjuntos derivados de MSigDB. Conjuntos de genes com P <0,05 foram considerados significativos. As pontuações do módulo de assinatura foram calculadas por meio da função "AddModuleScore", com configurações padrão em Seurat. Resumidamente, para cada célula, a pontuação foi definida como a expressão média da lista de genes de assinatura subtraindo a expressão média da lista de genes de controle correspondente. As listas de genes usadas para análise são fornecidas na Tabela Suplementar S11.
Análise Metacélula
Usamos o pacote R “MetaCell” 27 para analisar os dados. Removemos genes mitocondriais específicos, genes de imunoglobulina e genes ligados a modelos de transcrição com suporte insuficiente (anotados com o prefixo “Rp-“). Em seguida, filtramos células com menos de 500 UMIs. As características do gene foram selecionadas usando o parâmetro Tvm = 0,08 e uma contagem UMI total mínima> 100. Posteriormente, realizamos o agrupamento hierárquico da matriz de correlação entre esses genes (genes de filtragem com baixa cobertura e correlação de computação usando uma matriz UMI amostrada abaixo) e selecionados agrupamentos de genes contendo genes âncora. Usamos K = 100 e 500 iterações de bootstrap e outros parâmetros padrão. As metacélulas foram anotadas pelos tipos de células mais abundantes que compõem cada metacelete.
Análise de rede regulatória de genes
Para identificação e pontuação da atividade do regulon, empregamos o fluxo de trabalho pySCENIC28,29 em dados de metacélulas log-normalizadas para determinar conjuntos de genes coexpressos. Ligamos alvos diretos aos seus fatores de transcrição correspondentes usando bancos de dados RcisTarget (v1.2.1) e retemos genes a jusante putativos com motivos de DNA enriquecidos a 10 kb ou 500 bp do local de início da transcrição (pontuação de enriquecimento normalizado> 3). Finalmente, usamos a função AUCell para pontuar a atividade de cada regulon entre as células no conjunto de dados, que foi calculado como a soma dos genes expressos por regulon e produziu matrizes de atividade binária com base em pontos de corte ajustados manualmente após inspecionar as distribuições de pontuações AUC. Os escores de especificidade de regulagem (RSS) foram calculados pela função “regulon_specificity_scores” do algoritmo pySCENIC com parâmetros padrão.
Análise da resposta de IFN-α / β de PBMCs
PBMCs foram isolados de sangue heparinizado por Ficoll-Hypaque a 400 × g por 30 min. Os PBMCs (1 × 106 ml – 1) dos doadores antes e após a vacinação foram então semeados em placas de cultura de 48 poços com RPMI-1640 contendo 5% de reposição de soro nocaute e 0,032% de heparina. No dia seguinte, o meio foi trocado e as células foram tratadas com 100 ng / ml de IFN-α e 10 ng / ml de IFN-β por 12 h. Algumas células foram pré-tratadas com 250 ng / ml de RBD por 16 h, seguido por tratamento com IFN-α / β por 12 h. Após a lavagem e extração do RNA total, foi realizada PCR quantitativa em tempo real para detectar a expressão de genes associados à resposta ao interferon tipo I. As alterações de dobramento em relação a GAPDH foram calculadas por 2-ΔΔCt e expressas como médias ± SEM. As diferenças entre os grupos foram avaliadas pelo teste t de Student pareado e consideradas significativas quando P <0,05.
Análise estatística
Os dados clínicos foram resumidos pela média (desvio padrão), mediana (Q1, Q3) ou número (porcentagem), quando apropriado. O teste dos postos sinalizados de Wilcoxon foi usado para comparar medianas pareadas ao longo do tempo para características laboratoriais. Além disso, o teste de soma de Wilcoxon foi usado para comparar as alterações medianas da linha de base entre as coortes A e B. Classificamos os eventos adversos de acordo com a escala emitida pela China National Medical Products Administration (https://www.nmpa.gov. cn / xxgk / ggtg / qtggtg / 20191231111901460.html) e o julgamento dos resultados dos testes laboratoriais foi baseado no intervalo de valores de referência da população local. Todos os testes estatísticos foram bilaterais. Significância estatística foi definida como P ≤ 0,05. As análises estatísticas foram realizadas usando SAS v9.4 (SAS Institute Inc., Cary, NC, EUA).
Disponibilidade de dados
Os números de acesso para o sequenciamento de dados brutos e dados processados ??neste artigo são Genome Sequence Archive no BIG Data Center (GSA, Instituto de Genômica de Pequim, Academia Chinesa de Ciências): HRA001150.
Publicado em 07/11/2021 22h56
Artigo original:
Estudo original:
- https://www.ncbi.nlm.nih.gov/labs/pmc/articles/PMC8546144/
- https://dx.doi.org/10.1038/s41421-021-00329-3


