A computação molecular é uma área de estudo promissora que visa utilizar moléculas biológicas para criar dispositivos programáveis. Essa ideia foi introduzida pela primeira vez em meados da década de 1990 e desde então tem sido realizada por vários cientistas da computação e físicos em todo o mundo.
Pesquisadores da East China Normal University e da Shanghai Jiao Tong University desenvolveram recentemente redes neurais convolucionais moleculares (CNNs) baseadas em circuitos reguladores de DNA sintético. Sua abordagem, apresentada em um artigo publicado na Nature Machine Intelligence, supera alguns dos desafios normalmente encontrados ao criar redes neurais artificiais eficientes baseadas em componentes moleculares.
“A interseção da ciência da computação e da biologia molecular é um terreno fértil para uma ciência nova e empolgante, especialmente o design de sistemas inteligentes é um objetivo de longa data para os cientistas”, disse Hao Pei, um dos pesquisadores que realizaram o estudo, ao TechXplore. “Em comparação com o cérebro, a escala e o poder de computação das redes neurais de DNA desenvolvidas são severamente limitados, devido às limitações de tamanho da rede. O objetivo principal do nosso trabalho era aumentar o poder de computação dos circuitos de DNA, introduzindo um modelo de rede neural adequado para sistemas moleculares de DNA.”
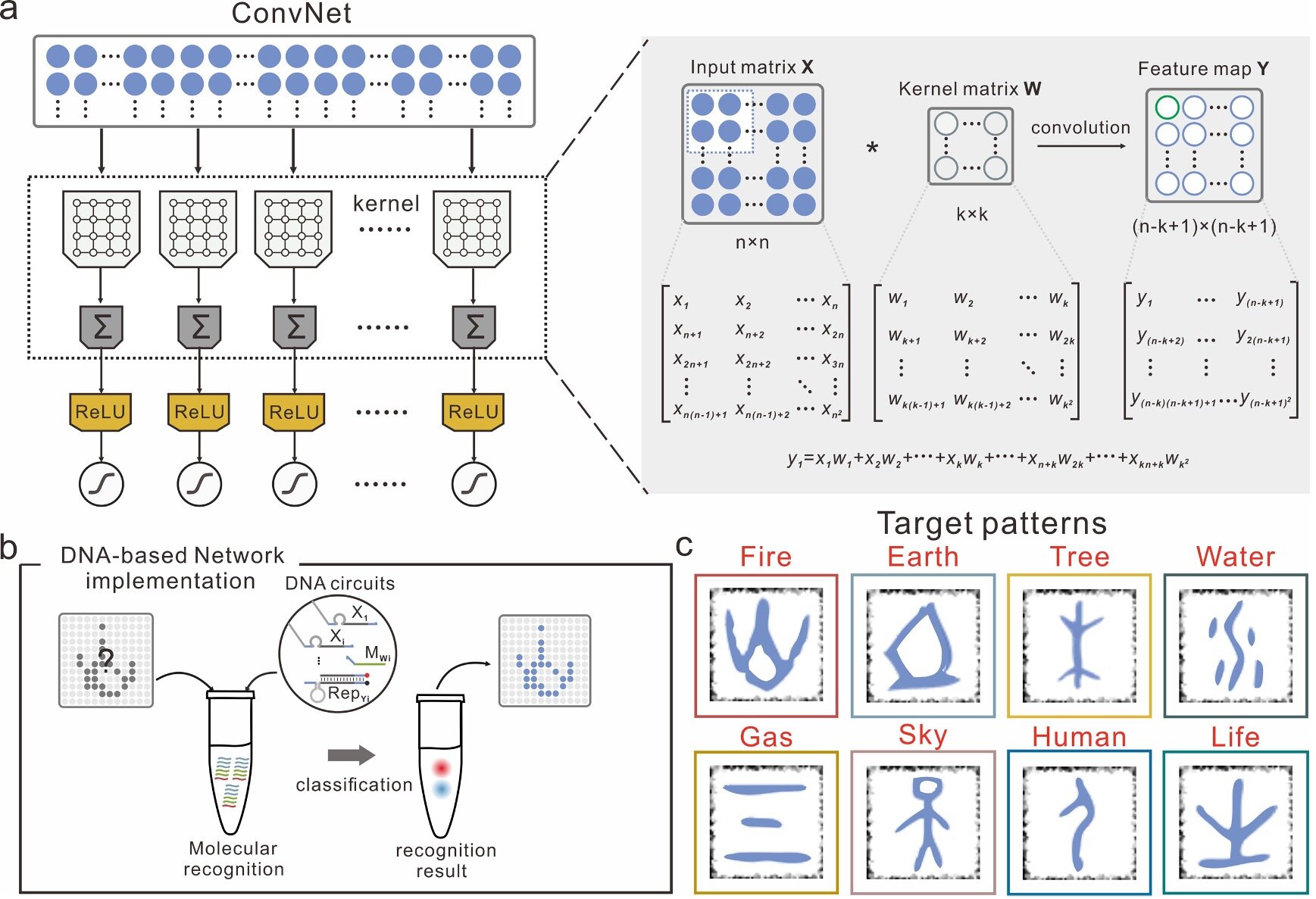
Ao realizar sua pesquisa, Pei e seus colegas descobriram que isso poderia ser particularmente promissor para modelar circuitos de DNA, devido à sua escassa conectividade topológica, que se assemelha à das redes neurais biológicas reais. Assim, eles decidiram usar CNNs para criar uma rede neural em larga escala baseada em DNA composta por 512 filamentos de DNA sintético. Notavelmente, sua rede proposta produz milhares de reações químicas e gera centenas de espécies moleculares.
“Nosso grupo se concentrou na engenharia e programação precisas de moléculas de ácidos nucleicos, e projetamos e construímos uma série de nanoestruturas dinâmicas de DNA que poderiam ser usadas como elementos reguladores para a construção de grandes circuitos”, explicou Pei. “Neste trabalho, usamos uma nanoestrutura de DNA dinâmica chamada de porta de comutação, que é funcionalmente semelhante aos riboswitches em circuitos reguladores de genes, todos consistindo em dois domínios funcionais independentes que detectam e respondem a entradas externas”.
A porta de comutação na rede dos pesquisadores permite que eles controlem independentemente suas funções de transmissão de sinal e funções de atribuição de peso por meio de um processo conhecido como comutação conformacional intramolecular. Este processo é particularmente adequado para o compartilhamento de peso e conectividade esparsa de CNNs.
Os circuitos de DNA operam dentro da rede dos pesquisadores, onde todas as unidades de computação estão prontas para responder às entradas. Uma vez que as entradas são alimentadas para a solução, fitas simples de DNA irão desencadear reações de deslocamento de fita em cascata em ordem.
Essas reações, impulsionadas pela energia livre de Gibbs ou entropia no sistema, geram sinais fluorescentes correspondentes. Os pesquisadores codificaram todos os padrões de teste usando um conjunto de fitas simples de DNA e cada um dos sinais de fluorescência gerados representa um desses padrões de teste.
“Nós estendemos o principal recurso da CNN – topologia esparsa e arquitetura de compartilhamento de peso para uma rede neural de DNA, que pode efetivamente reduzir a complexidade e os parâmetros da arquitetura de rede por meio de neurônios esparsamente conectados”, disse Pei. “Para implementar esses recursos, projetamos uma arquitetura de porta de comutação composta por dois domínios funcionais independentes (domínio de ajuste de peso e domínio de reconhecimento).”
A abordagem baseada em CNN tem várias vantagens sobre os métodos de computação molecular propostos anteriormente. Em primeiro lugar, sua arquitetura de porta de comutação pode ser usada para incorporar comutadores moleculares responsivos a ligantes. Isso permitiria que a rede adaptasse suas funções em resposta às mudanças ambientais, potencialmente possibilitando o desenvolvimento de circuitos moleculares que se assemelham a redes neurais biológicas e capazes de comportamento “inteligente”.
Além disso, a natureza inerentemente paralela das moléculas de DNA poderia permitir a paralelização autônoma das operações da CNN. Isso pode ser particularmente valioso para alcançar o processamento de informações escalável.
“Nós propusemos uma estratégia sistemática para implementar o algoritmo ConvNet no nível molecular”, disse Pei. “Sentimos que nosso método é um grande avanço em sistemas artificiais de processamento de informações moleculares, pois alcançou tarefas de classificação rápidas e precisas que poderiam classificar 32 padrões moleculares em 30 minutos, o que pode ser a computação química artificial mais rápida, poderosa e complexa sistema até à data, tanto quanto sabemos.”
O trabalho recente de Pei e seus colegas apresenta uma arquitetura alternativa baseada em DNA que pode informar o projeto de novos sistemas de computação molecular. No futuro, sua abordagem poderá ser usada para criar vários dispositivos de diagnóstico molecular para aplicações biomédicas.
“Ao fazer interface com entradas sensoriais, a ConvNet baseada em DNA poderia, em princípio, usar centenas de alvos como entradas e facilitar aplicações mais amplas em diagnóstico de doenças, perfis de padrões de expressão e medicina de precisão”, acrescentou Pei. “Com base neste modelo ConvNet baseado em DNA, agora planejamos construir classificador molecular que pode ser usado para classificação diagnóstica de várias doenças”.
Publicado em 28/07/2022 11h22
Artigo original:
Estudo original:


