Desde que o pioneiro da inteligência artificial, Marvin Minsky, patenteou o princípio da microscopia confocal em 1957, ela se tornou o burro de carga em laboratórios de ciências da vida em todo o mundo, devido ao seu contraste superior sobre a microscopia de campo amplo tradicional. No entanto, os microscópios confocais não são perfeitos. Eles aumentam a resolução ao obter imagens de apenas um único ponto em foco por vez, então pode demorar um pouco para digitalizar uma amostra biológica inteira delicada, expondo-a a pequenas doses que podem ser tóxicas.
Para levar a imagem confocal a um nível de desempenho sem precedentes, uma colaboração do Laboratório Biológico Marinho (MBL) inventou uma plataforma confocal “pia de cozinha” que pega emprestado soluções de outros sistemas de imagem de alta potência, adiciona um fio unificador de “Aprendizado profundo” algoritmos de inteligência artificial e melhora com sucesso a resolução volumétrica do confocal em mais de 10 vezes, reduzindo simultaneamente a fototoxicidade. Seu relatório sobre a tecnologia, chamado “Multiview Confocal Super-Resolution Microscopy”, foi publicado online esta semana na Nature.
“Muitos laboratórios têm confocais, e se eles podem extrair mais desempenho deles usando esses algoritmos de inteligência artificial, então eles não precisam investir em um microscópio totalmente novo. Para mim, esse é um dos melhores e mais empolgantes motivos para adotar esses métodos de IA “, disse o autor sênior e membro do MBL Hari Shroff do Instituto Nacional de Imagem Biomédica e Bioengenharia.
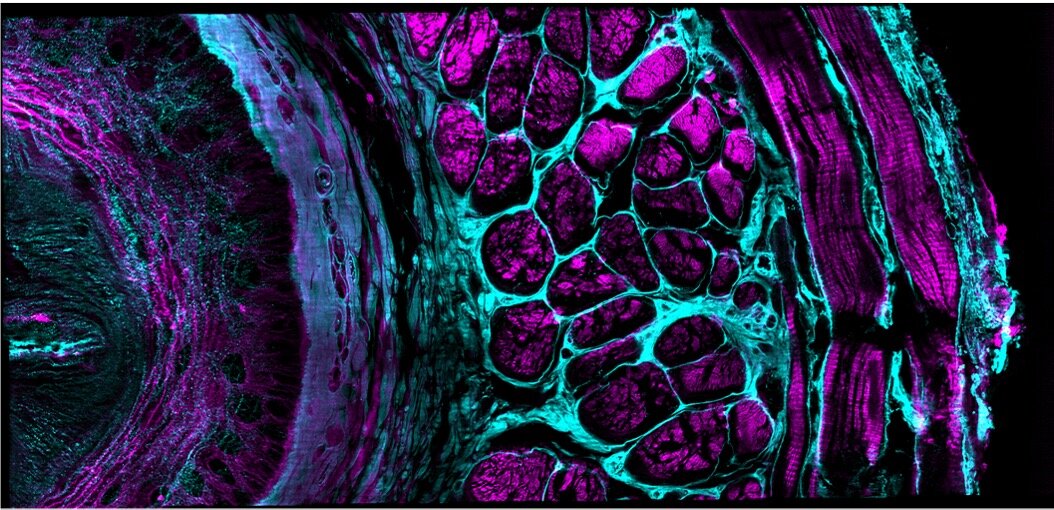
Entre suas inovações, a nova plataforma confocal usa três lentes objetivas, permitindo a visualização de uma ampla variedade de tamanhos de amostra, desde núcleos e neurônios no embrião de C. elegans até o verme adulto completo. Múltiplas visualizações de espécimes são rapidamente capturadas, registradas e fundidas para produzir reconstruções com resolução melhorada em comparação à microscopia confocal de visualização única. A plataforma também apresenta cabeças de leitura inovadoras para as três lentes, permitindo que a iluminação de varredura em linha seja facilmente adicionada à base do microscópio.
Além disso, a equipe adicionou capacidade de “super-resolução” à plataforma (resolução aprimorada além do limite de difração da luz), adaptando técnicas de microscopia de iluminação estruturada.
“O ápice do hardware que é escalado nesta plataforma são as múltiplas lentes ao redor da amostra, e então o truque da super-resolução, que requer uma combinação de hardware e computação para ser alcançado. É um tour de force, mas é uma receita bastante fototóxica. Há muita luz sendo entregue à amostra “, disse o co-autor e MBL Fellow Patrick La Rivière, da Universidade de Chicago.
Uma maneira de lidar com a fototoxicidade é diminuir a luz proveniente do laser do microscópio. Mas então você começa a ter problemas com “ruído” na imagem – granulação do fundo que pode obscurecer os detalhes do objeto que você deseja criar (o “sinal”). É aqui que entra a inteligência artificial.
A equipe treinou um modelo de computador Deep Learning, ou rede neural, para distinguir entre imagens de baixa qualidade com uma relação sinal-ruído (SNR) baixa e imagens melhores com um SNR mais alto. “Eventualmente, a rede poderia prever as imagens SNR mais altas, mesmo com uma entrada SNR bastante baixa”, disse Shroff.
“O Deep Learning permite que você tome este encontro de hardware como o padrão ouro para resolução e, em seguida, treine uma rede neural para obter resultados semelhantes com dados SNR muito mais baixos, muito menos aquisições e muito menos dose de luz para a amostra”, disse La Rivière .
A equipe demonstrou as capacidades da plataforma em mais de 20 amostras fixas e vivas diferentes, visando estruturas que variavam de menos de 100 nanômetros a um milímetro. Estes incluíram distribuições de proteínas em células individuais; núcleos e neurônios em desenvolvimento em embriões, larvas e adultos de C. elegans; mioblastos em discos imaginais da asa de Drosophila e tecidos renais, esofágicos, cardíacos e cerebrais de camundongos. Eles também veem aplicações potenciais para imagens de tecido humano em laboratórios de histologia e patologia.
Shroff, La Rivière e o co-autor e biólogo celular Daniel Colón-Ramos da Yale School of Medicine têm colaborado na MBL há quase uma década para desenvolver tecnologias de imagem com maior velocidade, resolução e maior duração. Colaboradores nesta plataforma confocal também incluíram a Applied Scientific Instrumentation, uma empresa com a qual trabalharam no MBL e no National Institutes of Health.
Yicong Wu, primeiro autor do artigo, construiu a nova plataforma confocal e implantou suas abordagens de Deep Learning. Wu aprendeu a usar Deep Learning no MBL na versão piloto de um novo curso lançado este ano, DL @ MBL: Deep Learning for Microscopy Image Analysis. (La Rivière é docente do curso.)
“É uma prova do curso que Yicong poderia aprender métodos de Aprendizado Profundo em 4 dias e inovar rapidamente com eles, então agora podemos aplicá-los em nosso laboratório”, disse Shroff. “Esse é um esquema de feedback curto, certo? Foi ótimo que o MBL o tenha catalisado.”
Publicado em 25/11/2021 09h12
Artigo original:
Estudo original:


