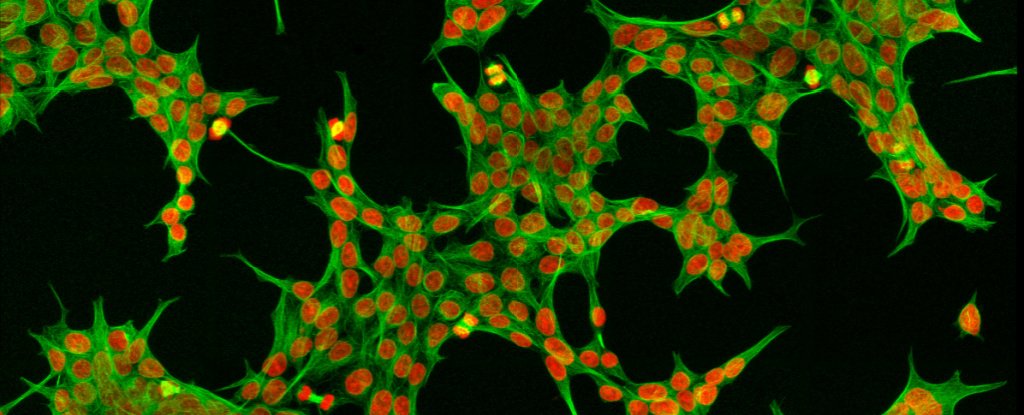
Dentro de cada célula do corpo humano existe uma constelação de proteínas, milhões delas. Eles estão todos se acotovelando, sendo rapidamente montados, dobrados, embalados, despachados, cortados e reciclados em uma colmeia de atividade que funciona em um ritmo febril para nos manter vivos e funcionando.
Mas sem um inventário completo do universo de proteínas dentro de nossas células, os cientistas são pressionados a avaliar em um nível molecular o que há de errado em nosso corpo que leva à doença.
Agora, os pesquisadores desenvolveram uma nova técnica que usa inteligência artificial para assimilar dados de imagens microscópicas de células individuais e análises bioquímicas, para criar um “mapa unificado” de componentes subcelulares – metade dos quais, ao que parece, nunca vimos antes .
“Os cientistas perceberam há muito tempo que há mais coisas que não sabemos do que sabemos, mas agora finalmente temos uma maneira de olhar mais a fundo”, diz o cientista da computação e biólogo de redes Trey Ideker, da Universidade da Califórnia (UC) em San Diego.
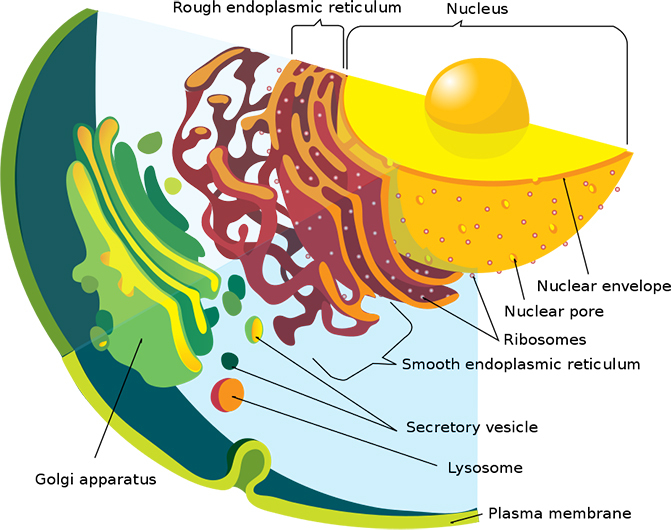
Os microscópios, por mais poderosos que sejam, permitem aos cientistas examinar o interior de células individuais, até o nível de organelas como as mitocôndrias, os pacotes de energia das células, e os ribossomos, as fábricas de proteínas. Podemos até adicionar corantes fluorescentes para marcar e rastrear proteínas facilmente.
As técnicas de bioquímica podem ser ainda mais profundas, concentrando-se em proteínas individuais usando, por exemplo, anticorpos direcionados que se ligam à proteína, puxam-na para fora da célula e ver o que mais está ligado a ela.
Integrar essas duas abordagens é um desafio para os biólogos celulares.
“Como você preenche essa lacuna do nanômetro para a escala de mícron? Esse tem sido um grande obstáculo nas ciências biológicas”, explica Ideker.
“Acontece que você pode fazer isso com inteligência artificial – ver dados de várias fontes e pedir ao sistema para montá-los em um modelo de célula.”
O resultado: Ideker e seus colegas inverteram mapas didáticos de células globulares que nos dão uma visão panorâmica de organelas coloridas em uma intrincada teia de interações proteína-proteína, organizadas por pequenas distâncias entre elas.
Combinando dados de imagem de uma biblioteca chamada Human Protein Atlas e mapas existentes de interações de proteínas, o algoritmo de aprendizado de máquina foi encarregado de calcular as distâncias entre os pares de proteínas.
O objetivo era identificar comunidades de proteínas, chamadas assemblies, que coexistem em células em diferentes escalas, desde as muito pequenas (menos de 50 nm) até as muito ‘grandes’ (mais de 1 ?m).
Uma tímida das 70 comunidades de proteínas foi classificada pelo algoritmo, que foi treinado usando uma biblioteca de referência de proteínas com diâmetros conhecidos ou estimados e validada com experimentos posteriores.
Cerca de metade dos componentes da proteína identificados são aparentemente desconhecidos da ciência, nunca documentados na literatura publicada, sugerem os pesquisadores.
Na mistura estava um grupo de proteínas formando uma estrutura desconhecida, que os pesquisadores descobriram ser provavelmente responsável por processar e cortar transcrições recém-feitas do código genético que são usadas para fazer proteínas.
Outras proteínas mapeadas incluíram sistemas de transporte transmembrana que bombeiam suprimentos para dentro e para fora das células, famílias de proteínas que ajudam a organizar cromossomos volumosos e complexos de proteínas cujo trabalho é produzir, digamos, mais proteínas.
Um esforço pesado, porém, não é a primeira vez que cientistas tentam mapear o funcionamento interno das células humanas.
Outros esforços para criar mapas de referência de interações de proteínas produziram números surpreendentes e tentaram medir os níveis de proteína nos tecidos do corpo humano.
Os pesquisadores também desenvolveram técnicas para visualizar e rastrear a interação e o movimento das proteínas nas células.
Este estudo piloto dá um passo adiante, aplicando aprendizado de máquina a imagens de microscopia celular que localizam proteínas em relação a grandes marcos celulares, como o núcleo, e dados de estudos de interação de proteínas que identificam os vizinhos mais próximos de uma proteína em nanoescala.
“A combinação dessas tecnologias é única e poderosa porque é a primeira vez que medições em escalas muito diferentes foram reunidas”, disse o bioinformático Yue Qin, também da UC San Diego.
Ao fazer isso, a técnica de célula integrada em escala múltipla ou MuSIC “aumenta a resolução da imagem enquanto dá às interações de proteínas uma dimensão espacial, abrindo caminho para incorporar diversos tipos de dados em mapas de células de todo o proteoma”, escrevem Qin, Ideker e colegas .
Para ficar claro, essa pesquisa é muito preliminar: a equipe se concentrou em validar seu método e apenas analisou os dados disponíveis de 661 proteínas em um tipo de célula, uma linhagem de células renais que os cientistas vêm cultivando em laboratório há cinco décadas.
Os pesquisadores planejam aplicar sua técnica ultramoderna a outros tipos de células, diz Ideker.
Mas, enquanto isso, teremos que aceitar humildemente que somos meros intrusos dentro de nossas próprias células, capazes de compreender uma pequena fração do proteoma total.
“Eventualmente, poderemos ser capazes de compreender melhor a base molecular de muitas doenças, comparando o que é diferente entre células saudáveis e doentes”, diz Ideker.
Publicado em 28/11/2021 01h12
Artigo original:
Estudo original:


