Os métodos de inteligência artificial estão se movendo para a pesquisa do câncer.
Enquanto as células cancerosas se espalham em um prato de cultura, Guillaume Jacquemet está observando. Os movimentos celulares fornecem pistas de como drogas ou variantes genéticas podem afetar a disseminação de tumores no corpo, e ele está rastreando o núcleo de cada célula quadro após quadro de filmes de microscopia de lapso de tempo. Mas, como ele gerou cerca de 500 filmes, cada um com 120 quadros e 200-300 células por quadro, essa análise é desafiadora para dizer o mínimo. “Se eu tivesse que fazer o rastreamento manualmente, seria impossível”, diz Jacquemet, biólogo celular da Universidade Åbo Akademi em Turku, Finlândia.
Então, ele treinou uma máquina para localizar os núcleos. Jacquemet usa métodos disponíveis em uma plataforma chamada ZeroCostDL4Mic, parte de uma coleção crescente de recursos que visam tornar a tecnologia de inteligência artificial (IA) acessível a cientistas de bancada com experiência mínima de codificação.
As tecnologias de IA abrangem vários métodos. Um, chamado aprendizado de máquina, usa dados que foram pré-processados manualmente e faz previsões de acordo com o que a IA aprende. O aprendizado profundo, por outro lado, pode identificar padrões complexos em dados brutos. É usado em carros autônomos, software de reconhecimento de voz, computadores de jogos – e para localizar núcleos de células em enormes conjuntos de dados de microscopia.
O aprendizado profundo tem suas origens na década de 1940, quando os cientistas construíram um modelo de computador organizado em camadas interconectadas, como os neurônios do cérebro humano. Décadas depois, os pesquisadores ensinaram essas “redes neurais” a reconhecer formas, palavras e números. Mas foi só há cerca de cinco anos que o aprendizado profundo começou a ganhar força na biologia e na medicina.
Uma grande força motriz tem sido o crescimento explosivo dos dados de ciências biológicas. Com as tecnologias modernas de sequenciamento de genes, um único experimento pode produzir gigabytes de informação. O Cancer Genome Atlas, lançado em 2006, coletou informações sobre dezenas de milhares de amostras abrangendo 33 tipos de câncer; os dados excedem 2,5 petabytes (1 petabyte equivale a 1 milhão de gigabytes). E os avanços na rotulagem de tecidos e microscopia automatizada estão gerando dados de imagem complexos mais rápido do que os pesquisadores podem minerá-los. “Definitivamente, há uma revolução acontecendo”, diz Emma Lundberg, bioengenheira do KTH Royal Institute of Technology em Estocolmo.
Impulso de perfil baseado em imagem
O biólogo do câncer Neil Carragher teve o primeiro vislumbre dessa revolução em 2004. Ele estava liderando uma equipe da AstraZeneca em Loughborough, no Reino Unido, que explora novas tecnologias para as ciências da vida, quando se deparou com um estudo que fez a empresa repensar seu rastreio de drogas esforços. Ele e sua equipe vinham usando telas baseadas em células para procurar candidatos a medicamentos promissores, mas era difícil encontrar resultados. O estudo sugeria que a IA e a análise poderiam ajudá-los a melhorar seus processos de triagem. “Achamos que essa poderia ser uma solução para a crise de produtividade” diz Carragher.
Mas as tecnologias de IA podem ser difíceis de dominar pelos biólogos. Jacquemet diz que uma vez passou mais de uma semana tentando instalar as bibliotecas de software corretas para executar um modelo de aprendizado profundo. Então, ele diz, “você precisa aprender a codificar em Python” para usá-lo.
A equipe da AstraZeneca de Carragher trabalhou com a bióloga computacional Anne Carpenter e seus colegas no Broad Institute of MIT e Harvard em Cambridge, Massachusetts, para ampliar o método de perfil de imagem usado no artigo de 2004 e para investigar os efeitos de vários medicamentos na mama humana. células cancerosas. Carpenter desenvolveu a técnica em um procedimento chamado Cell Painting, que tinge as células com um painel de corantes fluorescentes e, em seguida, usa o software de código aberto CellProfiler para gerar perfis das células.
Ainda assim, essas análises podem ser trabalhosas, diz Carragher, que agora chefia a descoberta de medicamentos para o câncer na Universidade de Edimburgo, no Reino Unido. Mesmo com ferramentas de código aberto que evitavam a necessidade de codificar o aprendizado de máquina do zero – e um cluster de computação com milhares de processadores e terabytes de memória – poderia levar um mês ou mais para descobrir quais recursos celulares eles deveriam contar à imagem- software de análise para olhar, Carragher diz. E depois de otimizar os parâmetros para cada linha celular, sua equipe teve que fazer mais ajustes para fazê-la funcionar em todas as células.
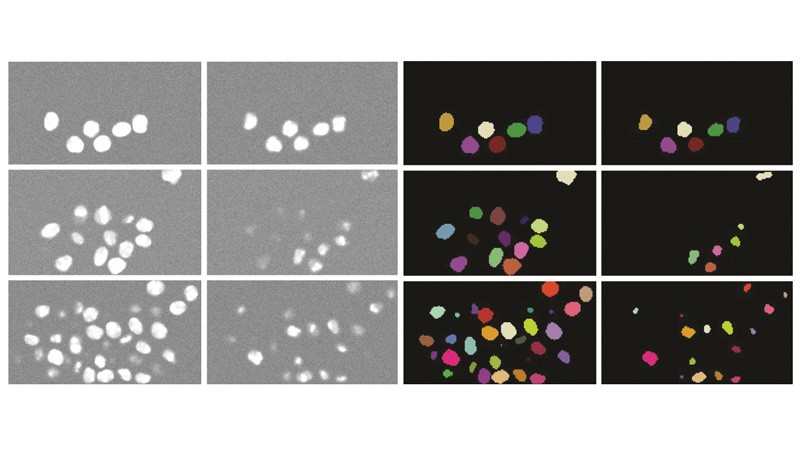
No ano passado, ele e sua equipe exploraram como o aprendizado profundo pode melhorar esse processo. O impulso foi uma análise de 2017 postada no servidor de pré-impressão bioRxiv por pesquisadores na sede do Google em Mountain View, Califórnia. Os pesquisadores baixaram o conjunto de dados de câncer de mama de Carragher da coleção Broad Bioimage Benchmark e o usaram para treinar uma rede neural profunda que antes tinha visto apenas imagens gerais, como carros e animais. Ao escanear padrões nos dados do câncer de mama, o modelo aprendeu a discernir as mudanças celulares que são significativas para a descoberta de medicamentos. Como o software não foi informado sobre o que procurar, ele encontrou recursos que os pesquisadores nem haviam considerado.
Com base nesse esforço, Carragher e seus colegas examinaram 14.000 compostos em 8 formas de câncer de mama. “Identificamos alguns resultados interessantes”, diz ele – incluindo um composto que já era conhecido por modular os receptores da serotonina, que é importante no desenvolvimento da glândula mamária, conforme relatado no início deste ano.
No Broad Institute, uma equipe liderada pelo biólogo computacional Juan Caicedo está aplicando perfis baseados em imagens para detectar mutações genéticas. Ele e sua equipe superexpressaram várias variantes de genes em células de câncer de pulmão, as coraram com o protocolo Cell Painting e procuraram por diferenças nas células que sugerissem possíveis oportunidades farmacêuticas. Eles descobriram que o aprendizado de máquina pode identificar variantes significativas em imagens, bem como processos que medem a expressão do gene nas células. Os pesquisadores relataram seus resultados na Conferência de Manufatura e Descoberta de Drogas Alimentadas pela AI em fevereiro no Instituto de Tecnologia de Massachusetts em Cambridge.
Como parte da Cancer Cell Map Initiative, que mapeia as redes moleculares subjacentes ao câncer humano, os pesquisadores estão treinando um modelo de aprendizado profundo para prever as respostas aos medicamentos com base na sequência do genoma do câncer de uma pessoa. Essas previsões têm implicações de vida ou morte, e a precisão é crucial, diz Trey Ideker, bioengenheiro da Universidade da Califórnia, em San Diego. Mas alguns relutam em aceitar os resultados quando os mecanismos por trás deles não são claros e as redes neurais profundas produzem respostas sem revelar seu processo – um problema conhecido como aprendizado de “caixa preta”. “Você quer saber por quê”, diz Ideker. “Você quer saber o mecanismo.” A equipe de Ideker está criando uma rede neural “visível”, que liga o funcionamento interno do modelo mais diretamente à biologia celular cancerígena. Como prova de conceito, a equipe criou um modelo para células de levedura. Chamado DCell, ele pode prever os efeitos das mutações gênicas no crescimento celular e nas vias moleculares subjacentes a esses efeitos.
A dimensão espacial
Lundberg e outros na Suécia estão usando o aprendizado profundo para enfrentar outro desafio computacional: avaliar a localização de proteínas. O trabalho faz parte do Human Protein Atlas, um esforço multianual e multimônico para mapear todas as proteínas humanas. A informação espacial revela onde as proteínas estão localizadas nas células e tendem a ser sub-representadas em estudos de nível de sistema, diz Lundberg. Mas se os pesquisadores soubessem dessa informação, eles poderiam usá-la para colher insights sobre a biologia subjacente, ela sugere.
Digite AI. Em 2016, Lundberg e seus colegas convidaram jogadores para ajudar os computadores a classificar a localização das proteínas nas células. Os cientistas cidadãos participaram de um jogo de RPG chamado EVE Online, no qual eles tinham que localizar proteínas marcadas com fluorescência para ganhar créditos de jogo, impulsionando um sistema de IA já usado para essa finalidade. Mas mesmo o sistema ampliado ficou atrás de especialistas humanos em termos de precisão e velocidade.
Então, em 2018, a equipe de Lundberg levou suas imagens para Kaggle – uma plataforma que desafia os especialistas em aprendizado de máquina a desenvolver seus melhores modelos para quebrar conjuntos de dados postados por empresas e pesquisadores. Ao longo de 3 meses, 2.172 equipes em todo o mundo competiram para desenvolver um modelo de aprendizado profundo que pudesse observar uma célula tingida por uma proteína e vários marcadores de referência e determinar a distribuição espacial da proteína.
A tarefa era desafiadora. Metade das proteínas humanas são encontradas em vários locais nas células, diz Lundberg. E alguns compartimentos celulares – o núcleo, por exemplo – são localizações muito mais comuns do que outros.
Ainda assim, os Kagglers cumpriram, diz Lundberg. A maioria das estratégias principais veio de cientistas computacionais sem formação em biologia – incluindo Bojan Tunguz, um engenheiro de software que criou modelos que prevêem terremotos e inadimplência de empréstimos antes de ganhar um dos primeiros lugares no concurso Human Protein Atlas. A abordagem desses problemas é semelhante em disciplinas muito diferentes, diz Tunguz.
O melhor modelo identificou locais raros e comuns em uma variedade de linhas de células e, o mais importante, captou bem os padrões mistos, diz Lundberg. O algoritmo funcionou quase tão precisamente quanto os especialistas humanos e com maior velocidade e reprodutibilidade. Além disso, poderia quantificar a informação espacial. “Quando podemos quantificá-lo, e não apenas descrevê-lo com um rótulo, podemos integrá-lo a outros tipos de dados.” Isso inclui dados ‘ômicos’, que já estão transformando a pesquisa sobre o câncer.
Uma estrutura computacional conhecida como DeepProg aplica aprendizado profundo a conjuntos de dados “ômicos”, incluindo expressão gênica e dados epigenéticos, para prever a sobrevivência do paciente, por exemplo. E o DigitalDLSorter prevê resultados inferindo tipos e quantidades de células imunológicas diretamente dos dados de sequenciamento de RNA de tumor, em vez de depender de laboriosos fluxos de trabalho convencionais.
No horizonte
Muitas das ferramentas necessárias para construir modelos de aprendizado profundo estão disponíveis gratuitamente online, incluindo bibliotecas de software e estruturas de codificação, como TensorFlow, Pytorch, Keras e Caffe. Os pesquisadores que desejam fazer perguntas e debater soluções para problemas que surgem com ferramentas de análise de imagens podem usar um recurso online chamado Scientific Community Image Forum. Também estão se tornando disponíveis repositórios que permitem aos pesquisadores encontrar e reaproveitar modelos de aprendizado profundo para tarefas relacionadas – um processo chamado aprendizado por transferência. Um exemplo é o Kipoi, que permite aos pesquisadores pesquisar e explorar mais de 2.000 modelos prontos para uso treinados para tarefas como prever como proteínas conhecidas como fatores de transcrição se ligarão ao DNA, ou onde as enzimas provavelmente irão emendar o código genético.
Trabalhando com outros desenvolvedores de ferramentas, a equipe de Lundberg montou um ‘zoológico modelo’ rudimentar (https://bioimage.io) para compartilhar rapidamente seus modelos de Atlas de Proteína Humana, e agora está criando um repositório mais sofisticado que será útil para produtores de modelos e usuários não especialistas.
Uma plataforma chamada ImJoy fará parte desse esforço, diz Lundberg. Criada por Wei Ouyang, uma pós-doc em seu laboratório, a plataforma permite que os pesquisadores testem e executem modelos de IA por meio de um navegador da web em seu computador, na nuvem ou em um telefone. O compartilhamento de conjuntos de dados de bioimagem e modelos de aprendizagem profunda também será uma prioridade para o Center for Open Bioimage Analysis, um esforço financiado pelo governo dos Estados Unidos e liderado por Carpenter e Kevin Eliceiri, um bioengenheiro da Universidade de Wisconsin – Madison.
Outra opção, ZeroCostDL4Mic, lançado no mês passado. Desenvolvido pelo biofísico Ricardo Henriques da University College London, ZeroCostDL4Mic usa Colab, o serviço de nuvem gratuito do Google para desenvolvedores de IA, para fornecer acesso a várias ferramentas populares de microscopia de aprendizado profundo, incluindo aquela que Jacquemet usa para automatizar a marcação de núcleos celulares em seus filmes. “Tudo o que você precisa é instalado em alguns minutos”, explica Jacquemet. Com alguns cliques do mouse, os usuários podem usar dados de exemplo para treinar uma rede neural para concluir a tarefa desejada (consulte “Procurado: mais dados”) e, em seguida, aplicar essa rede aos seus próprios dados – tudo sem a necessidade de código.
Os pesquisadores que desejam usar conjuntos de dados maiores ou treinar modelos mais complexos podem precisar comprar ou acessar recursos computacionais extras além do serviço gratuito do Google.
Ao facilitar o caminho para biólogos com escasso conhecimento e recursos para usar o aprendizado profundo, diz Henriques, ZeroCostDL4Mic atua como “uma droga de porta de entrada” para IA, atraindo pesquisadores para explorar o software subjacente a essas ferramentas que continuarão a transformar a pesquisa sobre câncer e além.
Publicado em 02/04/2021 19h58
Artigo original:
Estudo original:


