Graças ao advento dos antibióticos, a sífilis não é a mesma ameaça mortal de séculos atrás, mas ainda é uma infecção sexualmente transmissível comum em todo o mundo e que pode ser muito perigosa se não for tratada.
As estimativas apontam para novas infecções em todo o mundo em cerca de 6 milhões de casos por ano, com a doença causando mais de 100.000 mortes entre adultos a cada ano, embora o número de casos fetais e neonatais, expostos no útero, seja muito maior, com uma estimativa de 300.000 mortes anualmente.
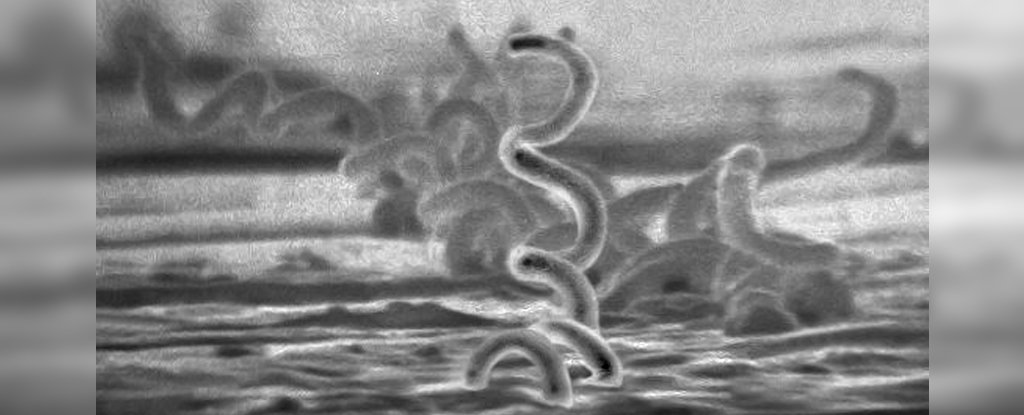
Apesar desse ônus global contínuo, a bactéria em forma de espiral que causa a sífilis – Treponema pallidum subespécie pallidum (daqui em diante T. pallidum) – continua sendo um mistério científico.
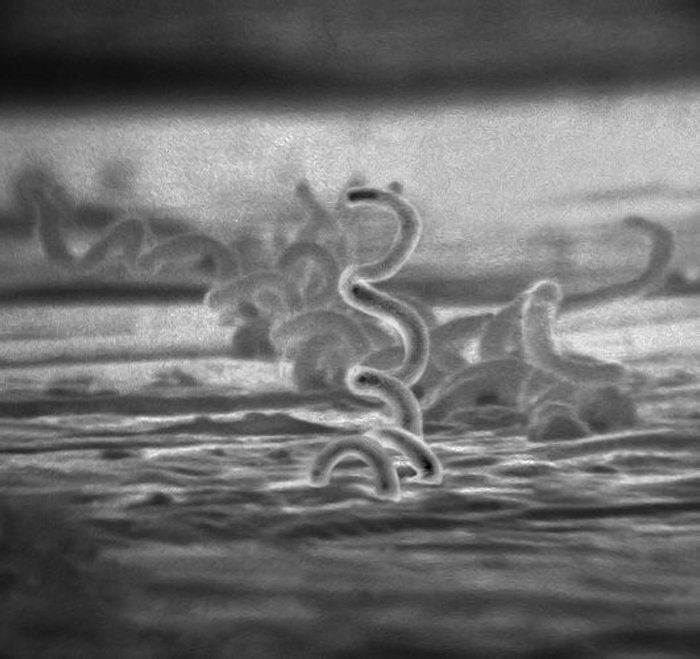
Esse patógeno helicoidal é um organismo particularmente evasivo, tanto no contexto de infecções – onde pode iludir o sistema imunológico (que dura algumas décadas dentro do corpo em alguns casos) – quanto em nossa capacidade de estudá-lo. Os cientistas aprenderam apenas recentemente como cultivar a entidade, após um longo século de tentativas.
No primeiro contexto, as formidáveis habilidades de T. pallidum como um ‘patógeno furtivo’ foram atribuídas à capa costurada por proteínas de sua superfície bacteriana, que parece escondê-la da detecção do sistema imunológico.
“A chave da capacidade do espiroqueta da sífilis para evasão imunológica e, portanto, a ‘patogenicidade furtiva’ é sua membrana externa incomum”, observou um grupo de pesquisadores em 2016.
Agora, uma nova investigação pode ser capaz de explicar como esse mecanismo furtivo é tão eficaz.
Cientistas da Universidade de Washington compararam as seqüências genômicas de cepas de T. pallidum retiradas de um homem que foi diagnosticado com sífilis quatro vezes ao longo de seis anos.
Comparando as cepas responsáveis pela primeira e pela última infecção, os pesquisadores queriam ver como as mudanças nos genes bacterianos podem ter aumentado a capacidade do T. pallidum de reinfetar esse paciente pobre.
Embora eles identificassem diferenças entre os dois genomas, as seqüências genéticas eram praticamente idênticas, refletindo uma “escassez de alterações de codificação no genoma” fora de um gene em particular.
Esse gene, chamado gene de repetição Treponema pallidum K (tprK), codifica uma das proteínas de superfície da bactéria, conhecida como TprK, e era muito diferente entre os dois genomas.
“Nas cerca de 1,1 milhão de bases que compõem o genoma da bactéria, houve cerca de 20 alterações no total. Isso é muito baixo”, diz o pesquisador chefe e patologista clínico Alex Greninger.
“Mas neste único gene, vimos centenas de mudanças”.
Os pesquisadores especulam que as mutações resultantes nas proteínas da superfície da TprK devido aos novos genes da tprK podem representar uma “base genética através da qual T. pallidum escapa da imunidade de proteção cruzada”, permitindo que a bactéria efetivamente se disfarce, alterando o arranjo de seus genes. membrana externa.
A equipe reconhece que suas descobertas exigem replicação adicional. Para começar, estamos analisando apenas o tamanho da amostra de um paciente aqui, e os pesquisadores também reconhecem as limitações inerentes às novas técnicas usadas para cultivar T. pallidum, que depende de modelos de coelho.
No entanto, certamente parece que eles descobriram algo estranho acontecendo aqui.
“Eu observei muitos genomas bacterianos, e eles são muito mais interessantes que os do Treponema, exceto esse gene”, explica o cientista Amin Addetia.
“Ele pode gerar um número impressionante de diversas seqüências dentro dessas regiões variáveis, sem prejudicar a capacidade da proteína de funcionar”.
Publicado em 01/05/2020 18h10
Artigo original:
Estudo original:
Achou importante? Compartilhe!
Assine nossa newsletter e fique informado sobre Astrofísica, Biofísica, Geofísica e outras áreas. Preencha seu e-mail no espaço abaixo e clique em “OK”:




