Nos cromossomos humanos, o DNA é revestido por proteínas para formar um cordão de contas extremamente longo. Essa “corda” é dobrada em várias alças, que se acredita ajudar as células a controlar a expressão gênica e facilitar o reparo do DNA, entre outras funções. Um novo estudo do MIT sugere que esses loops são muito dinâmicos e de vida mais curta do que se pensava anteriormente.
No novo estudo, os pesquisadores conseguiram monitorar o movimento de um trecho do genoma em uma célula viva por cerca de duas horas. Eles viram que esse trecho foi totalmente enrolado por apenas 3 a 6 por cento do tempo, com o loop durando apenas cerca de 10 a 30 minutos. As descobertas sugerem que a compreensão atual dos cientistas de como as alças influenciam a expressão gênica pode precisar ser revisada, dizem os pesquisadores.
“Muitos modelos no campo foram essas imagens de loops estáticos que regulam esses processos. O que nosso novo artigo mostra é que essa imagem não está realmente correta”, diz Anders Sejr Hansen, Professor Assistente de Desenvolvimento de Carreira Underwood-Prescott de Engenharia Biológica no MIT . “Sugerimos que o estado funcional desses domínios seja muito mais dinâmico.”
Hansen é um dos principais autores do novo estudo, juntamente com Leonid Mirny, professor do Instituto de Engenharia e Ciência Médica do MIT e do Departamento de Física, e Christoph Zechner, líder de grupo do Instituto Max Planck de Biologia Celular Molecular e Genetics em Dresden, Alemanha, e o Centro de Biologia de Sistemas Dresden. MIT pós-doc Michele Gabriele, recente Harvard University Ph.D. O ganhador Hugo Brandão e o estudante de pós-graduação do MIT Simon Grosse-Holz são os principais autores do artigo, que aparece hoje na Science.
Fora do laço
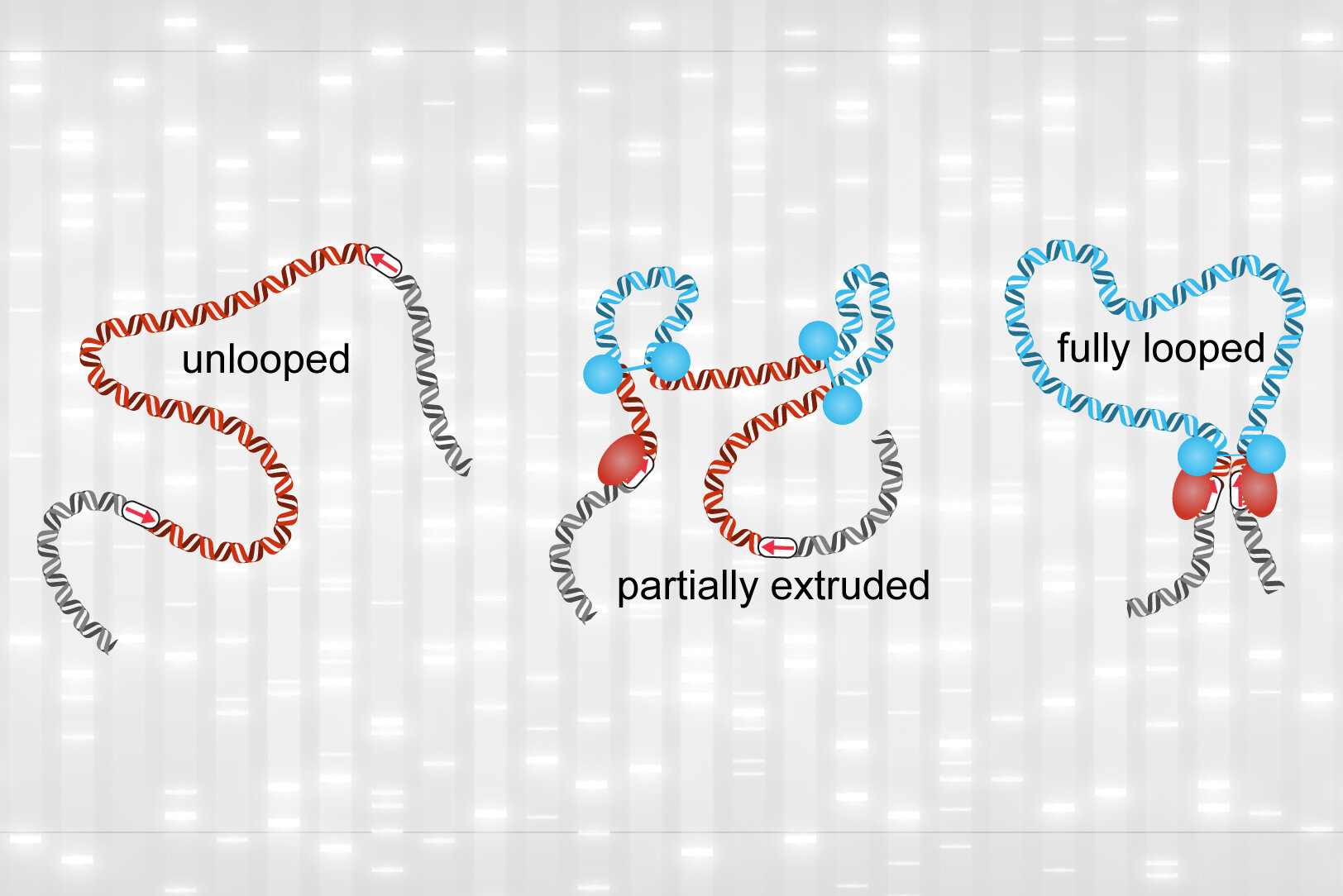
Usando simulações de computador e dados experimentais, cientistas, incluindo o grupo de Mirny no MIT, mostraram que os loops no genoma são formados por um processo chamado extrusão, no qual um motor molecular promove o crescimento de loops progressivamente maiores. O motor pára cada vez que encontra um “sinal de pare” no DNA. O motor que expulsa essas alças é um complexo proteico chamado coesina, enquanto a proteína ligada ao DNA CTCF serve como sinal de parada. Essas alças mediadas por coesina entre os locais CTCF foram observadas em experimentos anteriores.
No entanto, esses experimentos ofereceram apenas um instantâneo de um momento no tempo, sem informações sobre como os loops mudam ao longo do tempo. Em seu novo estudo, os pesquisadores desenvolveram técnicas que lhes permitiram marcar com fluorescência os locais de DNA CTCF para que pudessem visualizar os loops de DNA durante várias horas. Eles também criaram um novo método computacional que pode inferir os eventos de loop dos dados de imagem.
“Esse método foi crucial para distinguirmos sinal de ruído em nossos dados experimentais e quantificar o looping”, diz Zechner. “Acreditamos que essas abordagens se tornarão cada vez mais importantes para a biologia à medida que continuamos a aumentar os limites da detecção com experimentos”.
Os pesquisadores usaram seu método para obter imagens de um trecho do genoma em células-tronco embrionárias de camundongos. “Se colocarmos nossos dados no contexto de um ciclo de divisão celular, que dura cerca de 12 horas, o loop totalmente formado só existe por cerca de 20 a 45 minutos, ou cerca de 3 a 6 por cento do tempo”, diz Grosse-Holz. .
“Se o loop estiver presente apenas por um período tão pequeno do ciclo celular e de vida muito curta, não devemos pensar nesse estado totalmente em loop como sendo o regulador primário da expressão gênica”, diz Hansen. “Achamos que precisamos de novos modelos de como a estrutura 3D do genoma regula a expressão gênica, reparo de DNA e outros processos funcionais a jusante”.
Embora os loops totalmente formados fossem raros, os pesquisadores descobriram que os loops parcialmente extrudados estavam presentes em cerca de 92% das vezes. Esses loops menores foram difíceis de observar com os métodos anteriores de detecção de loops no genoma.
“Neste estudo, integrando nossos dados experimentais com simulações de polímeros, agora conseguimos quantificar as extensões relativas dos estados sem loop, parcialmente extrudado e totalmente em loop”, diz Brandão.
“Como essas interações são muito curtas, mas muito frequentes, as metodologias anteriores não conseguiam capturar totalmente sua dinâmica”, acrescenta Gabriele. “Com nossa nova técnica, podemos começar a resolver transições entre estados totalmente em loop e sem loop.”
Os pesquisadores levantam a hipótese de que esses loops parciais podem desempenhar papéis mais importantes na regulação do gene do que os loops totalmente formados. As fitas de DNA correm umas nas outras à medida que as alças começam a se formar e depois se desfazem, e essas interações podem ajudar elementos reguladores, como intensificadores e promotores de genes, a se encontrarem.
“Mais de 90 por cento do tempo, existem alguns loops transitórios, e presumivelmente o importante é ter esses loops que estão sendo extrudados perpetuamente”, diz Mirny. “O processo de extrusão em si pode ser mais importante do que o estado totalmente em loop que ocorre apenas por um curto período de tempo.”
Mais loops para estudar
Como a maioria das outras alças do genoma é mais fraca do que a que os pesquisadores estudaram neste artigo, eles suspeitam que muitas outras alças também se mostrarão altamente transitórias. Eles agora planejam usar sua nova técnica para estudar alguns desses outros loops, em uma variedade de tipos de células.
“Existem cerca de 10.000 desses loops, e analisamos um”, diz Hansen. “Temos muitas evidências indiretas para sugerir que os resultados seriam generalizáveis, mas não demonstramos isso. Usando a plataforma de tecnologia que montamos, que combina novos métodos experimentais e computacionais, podemos começar a abordar outros loops no genoma.”
Os pesquisadores também planejam investigar o papel de alças específicas na doença. Muitas doenças, incluindo um distúrbio do neurodesenvolvimento chamado síndrome FOXG1, podem estar ligadas à dinâmica defeituosa do loop. Os pesquisadores estão agora estudando como a forma normal e mutante do gene FOXG1, bem como o gene causador de câncer MYC, são afetados pela formação do loop do genoma.
Publicado em 18/04/2022 05h20
Artigo original:
Estudo original:


