
Enormes avanços na inteligência artificial significam que os pesquisadores podem projetar moléculas completamente originais em segundos, em vez de meses.
Em junho, os reguladores sul-coreanos autorizaram o primeiro medicamento, uma vacina COVID-19, sendo feita a partir de uma nova proteína projetada por humanos. A vacina é baseada em uma “nanopartícula” de proteína esférica que foi criada por pesquisadores há quase uma década, por meio de um processo de tentativa e erro de trabalho intensivo.
Agora, graças a avanços gigantescos em inteligência artificial (IA), uma equipe liderada por David Baker, bioquímico da Universidade de Washington (UW) em Seattle, relata na Science, que pode projetar tais moléculas em segundos em vez de meses.
Esses esforços fazem parte de uma mudança científica no mar, já que ferramentas de IA, como o software de previsão de estrutura de proteínas da DeepMind, AlphaFold, são adotadas por cientistas da vida. Em julho, a DeepMind revelou que a versão mais recente do AlphaFold havia previsto estruturas para todas as proteínas conhecidas pela ciência. E os últimos meses viram um crescimento explosivo nas ferramentas de IA – algumas baseadas no AlphaFold – que podem rapidamente criar proteínas completamente novas. Anteriormente, essa era uma busca meticulosa com altas taxas de falha.
“Desde o AlphaFold, houve uma mudança na maneira como trabalhamos com o design de proteínas”, diz Noelia Ferruz, bióloga computacional da Universidade de Girona, Espanha. “Estamos vivendo tempos muito emocionantes.”
A maioria dos esforços está focada em ferramentas que podem ajudar a produzir proteínas originais, com formas diferentes de qualquer coisa na natureza, sem muito foco no que essas moléculas podem fazer. Mas os pesquisadores – e um número crescente de empresas que estão aplicando IA ao design de proteínas – gostariam de projetar proteínas que possam fazer coisas úteis, desde a limpeza de resíduos tóxicos até o tratamento de doenças. Entre as empresas que estão trabalhando para esse objetivo estão a DeepMind em Londres e a Meta (anteriormente Facebook) em Menlo Park, Califórnia.
“Os métodos já são realmente poderosos. Eles vão ficar mais poderosos”, diz Baker. “A questão é quais problemas você vai resolver com eles.”
Do princípio
O laboratório de Baker passou as últimas três décadas produzindo novas proteínas. Um software chamado Rosetta, que seu laboratório começou a desenvolver na década de 1990, divide o processo em etapas. Inicialmente, os pesquisadores conceberam uma forma para uma nova proteína – muitas vezes juntando pedaços de outras proteínas – e o software deduziu uma sequência de aminoácidos que correspondia a essa forma.
Mas essas proteínas de “primeiro rascunho” raramente se dobravam na forma desejada quando feitas em laboratório e, em vez disso, acabavam presas em diferentes confirmações. Então, outro passo foi necessário para ajustar a sequência da proteína de forma que ela se dobrasse apenas em uma única estrutura desejada. Essa etapa, que envolvia simular todas as maneiras pelas quais diferentes sequências poderiam se dobrar, era computacionalmente cara, diz Sergey Ovchinnikov, biólogo evolucionário da Universidade de Harvard em Cambridge, Massachusetts, que costumava trabalhar no laboratório de Baker. “Você teria literalmente, tipo, 10.000 computadores rodando por semanas fazendo isso.”
Ao ajustar o AlphaFold e outros programas de IA, essa etapa demorada se tornou instantânea, diz Ovchinnikov. Em uma abordagem desenvolvida pela equipe de Baker, chamada alucinação, os pesquisadores alimentam sequências aleatórias de aminoácidos em uma rede de previsão de estrutura; isso altera a estrutura para que ela se torne cada vez mais parecida com uma proteína, conforme julgado pelas previsões da rede. Em um artigo de 2021, a equipe de Baker criou mais de 100 pequenas proteínas ‘alucinadas’ no laboratório e encontrou sinais de que cerca de um quinto se assemelhava à forma prevista.
AlphaFold e uma ferramenta semelhante desenvolvida pelo laboratório de Baker chamada RoseTTAFold, foram treinados para prever a estrutura de cadeias de proteínas individuais. Mas os pesquisadores logo descobriram que essas redes também poderiam modelar conjuntos de várias proteínas que interagem. Com base nisso, Baker e sua equipe estavam confiantes de que poderiam alucinar proteínas que se automontariam em nanopartículas de diferentes formas e tamanhos; estes seriam compostos por várias cópias de uma única proteína e seriam semelhantes àquelas em que a vacina COVID-19 se baseia.
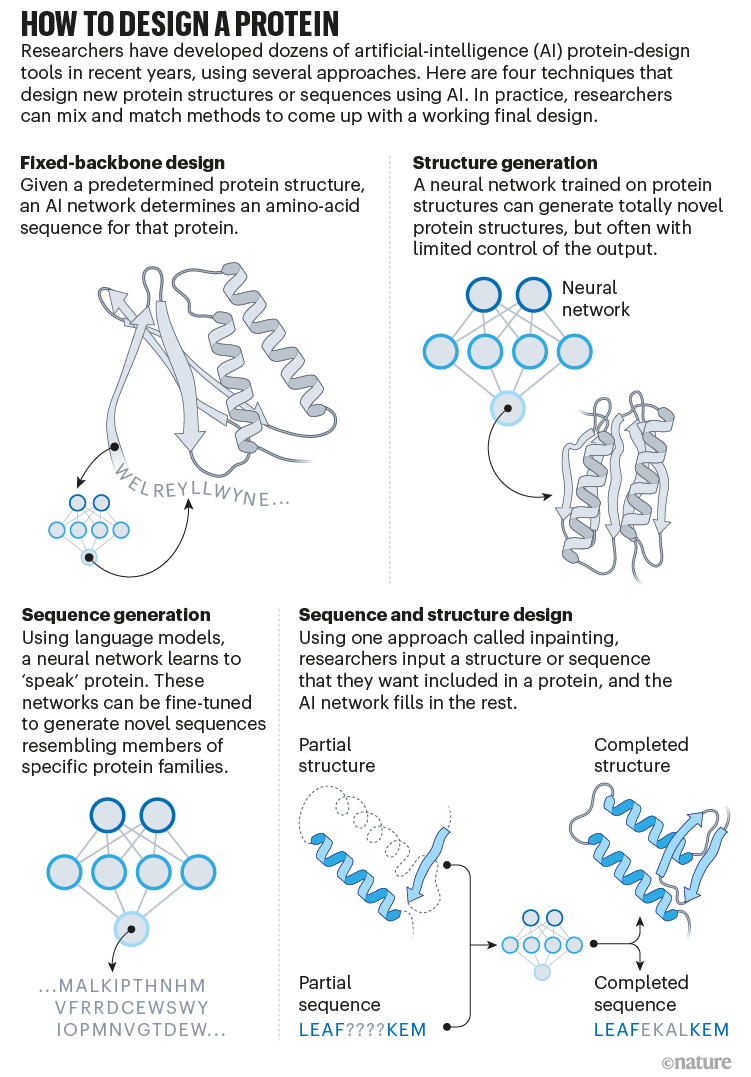
Mas quando eles instruíram os microrganismos fazendo suas criações nos laboratórios, nenhum dos 150 designs funcionou. “Eles não dobraram: eram apenas gosma no fundo do tubo de ensaio”, diz Baker.
Na mesma época, outro pesquisador do laboratório, o cientista de machine learning Justas Dauparas, estava desenvolvendo uma ferramenta de deep learning para resolver o que é conhecido como o problema de dobramento inverso – determinar uma sequência de proteína que corresponde à forma geral de uma determinada proteína. A rede, chamada ProteinMPNN, pode atuar como uma “verificação ortográfica” para proteínas de design criadas usando AlphaFold e outras ferramentas, diz Ovchinnikov, ajustando sequências enquanto mantém a forma geral das moléculas.
Quando Baker e sua equipe aplicaram essa segunda rede às suas nanopartículas de proteínas alucinadas, tiveram muito mais sucesso fazendo as moléculas experimentalmente. Os pesquisadores determinaram a estrutura de 30 de suas novas proteínas usando microscopia crioeletrônica e outras técnicas experimentais, e 27 delas combinaram com os projetos conduzidos por IA2. As criações da equipe incluíam anéis gigantes com simetrias complexas, diferentes de tudo encontrado na natureza. Em teoria, a abordagem poderia ser usada para projetar nanopartículas correspondentes a quase qualquer forma simétrica, diz Lukas Milles, biofísico que co-liderou o esforço. “É eletrizante ver o que essas redes podem fazer.”
Revolução do deep learning
Ferramentas de deep learning, como o proteinMPNN, mudaram o jogo no design de proteínas, diz Arne Elofsson, biólogo computacional da Universidade de Estocolmo. “Você extrai sua proteína, aperta um botão e obtém algo que uma em cada dez vezes funciona.” Taxas de sucesso ainda mais altas podem ser alcançadas combinando várias redes neurais para lidar com diferentes partes do processo de design, como a equipe de Baker fez ao projetar as nanopartículas. “Agora temos controle total sobre a forma da proteína”, diz Ovchinnikov.
A Baker’s não é o único laboratório que aplica IA ao design de proteínas. Em um artigo de revisão publicado no bioRxiv este mês, Ferruz e seus colegas contaram mais de 40 ferramentas de design de proteínas de IA que foram desenvolvidas nos últimos anos, usando várias abordagens (consulte ‘Como projetar uma proteína’).
Muitas dessas ferramentas, incluindo proteinMPNN, abordam o problema do dobramento inverso: elas especificam uma sequência que corresponde a uma estrutura específica, muitas vezes usando abordagens emprestadas de ferramentas de reconhecimento de imagem. Alguns outros são baseados em uma arquitetura semelhante à das redes neurais de linguagem, como GPT-3, que produz texto semelhante ao humano; mas, em vez disso, as ferramentas são capazes de produzir novas sequências de proteínas. “Essas redes são capazes de ‘falar’ proteínas”, diz Ferruz, que co-desenvolveu uma dessas redes.
Com tantas ferramentas de design de proteínas disponíveis, nem sempre fica claro a melhor forma de compará-las, diz Chloe Hsu, pesquisadora de machine learning da Universidade da Califórnia, Berkeley, que desenvolveu uma rede de dobramento inverso com pesquisadores da Meta.
Muitas equipes avaliam a capacidade de sua rede de determinar com precisão a sequência de uma proteína existente a partir de sua estrutura. Mas isso não se aplica a todos os métodos e não está claro como essa métrica, conhecida como taxa de recuperação, se aplica ao design de novas proteínas, dizem os cientistas. Ferruz gostaria de ver uma competição de design de proteínas, análoga ao experimento bienal Avaliação Crítica da Predição da Estrutura de Proteínas (CASP), no qual o AlphaFold demonstrou pela primeira vez sua superioridade sobre outras redes. “É um sonho. Algo como o CASP realmente faria o campo avançar”, diz ela.
Para o teste prático
Baker e seus colegas estão convencidos de que fazer uma nova proteína em laboratório é o teste final de seus métodos. Sua falha inicial em fazer montagens de proteínas alucinadas mostra isso. “O AlphaFold achava que eram proteínas fantásticas, mas claramente não funcionavam no laboratório úmido”, diz Basile Wicky, biofísico do laboratório de Baker que co-liderou o esforço, junto com Baker, Milles e o bioquímico da UW Alexis Courbet.
Mas nem todos os cientistas que desenvolvem ferramentas de IA para design de proteínas têm acesso fácil a configurações experimentais, observa Jinbo Xu, biólogo computacional do Instituto Tecnológico Toyota de Chicago, Illinois. Encontrar um laboratório para colaborar pode levar tempo, então Xu está estabelecendo seu próprio laboratório úmido para testar as criações de sua equipe.
Os experimentos também serão essenciais quando se trata de projetar proteínas com tarefas específicas em mente, diz Baker. Em julho, sua equipe descreveu um par de métodos de IA que permitem aos pesquisadores incorporar uma sequência ou estrutura específica em uma nova proteína. Eles usaram essas abordagens para projetar enzimas que catalisam reações específicas; proteínas capazes de se ligar a outras moléculas; e uma proteína que poderia ser usada em uma vacina contra um vírus respiratório que é uma das principais causas de hospitalização infantil.
No ano passado, a DeepMind lançou uma empresa spin-off chamada Isomorphic Labs em Londres, que pretende aplicar ferramentas de IA, como o AlphaFold, à descoberta de medicamentos. O executivo-chefe da DeepMind, Demis Hassabis, diz que vê o design de proteínas como uma aplicação óbvia e promissora para a tecnologia de deep learning e para o AlphaFold em particular. “Estamos trabalhando bastante no espaço de design de proteínas. É muito cedo.”
Publicado em 17/09/2022 10h57
Artigo original:
Estudo original:


