
Uma nova análise traça a história do impacto do rascunho do genoma na genômica desde 2001, vinculando seus efeitos em publicações, aprovações de medicamentos e compreensão da doença.
O 20º aniversário da publicação do primeiro esboço do genoma humano oferece uma oportunidade de rastrear como o projeto fortaleceu a pesquisa sobre as raízes genéticas das doenças humanas, mudou a descoberta de medicamentos e ajudou a revisar a ideia do próprio gene.
Aqui nós destilamos esses impactos e tendências. Combinamos vários conjuntos de dados para quantificar os diferentes tipos de elementos genéticos que foram descobertos e que geraram publicações, e como o padrão de descoberta e publicação mudou ao longo dos anos. Nossa análise uniu dados, incluindo 38.546 transcrições de RNA; cerca de 1 milhão de polimorfismos de nucleotídeo único (SNPs); 1.660 doenças humanas com raízes genéticas documentadas; 7.712 produtos farmacêuticos aprovados e experimentais; e 704.515 publicações científicas entre 1900 e 2017 (ver Informações complementares; SI).
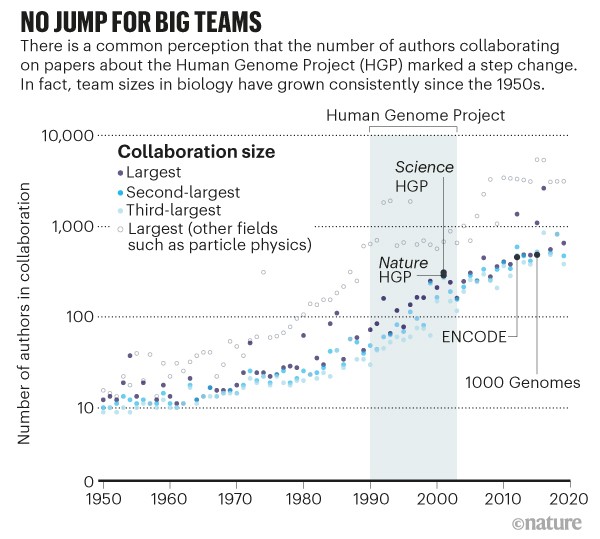
Os resultados destacam como o Projeto Genoma Humano (HGP), com sua lista abrangente de genes codificadores de proteínas, estimulou uma nova era de elucidação da função da porção não codificadora do genoma e abriu o caminho para desenvolvimentos terapêuticos. Crucialmente, os resultados rastreiam o surgimento de uma visão em nível de sistema da biologia ao lado da perspectiva convencional de um único gene, conforme os pesquisadores mapearam as interações entre os blocos de construção celulares (consulte ?Sem salto para grandes equipes?).
Existem limitações para nossa análise. Por exemplo, não há consenso sobre onde um gene começa e termina ou, surpreendentemente, até mesmo qual sequência codifica exatamente alguns genes. Várias convenções de nomenclatura são usadas para alguns elementos genômicos, portanto, às vezes, nossa metodologia não os conectava. E outros links entre publicações e elementos podem não ter sido adicionados às bases de dados pelos autores. Finalmente, nossos gráficos terminam em 2017, porque pode haver um lapso de tempo entre a publicação de um artigo e a entrada nas bases de dados que usamos.
No entanto, não esperamos que esses problemas afetem as tendências que observamos em como a pesquisa do genoma mudou ao longo do tempo. As tendências permanecem quando controlamos o crescimento das publicações de biologia no mesmo período (ver SI, Fig. S6). Não controlamos o tempo desde a descoberta dos genes, mas estimamos que fazer isso não teria alterado nossas conclusões.
Essas conexões oferecem um instantâneo da evolução do cenário de pesquisa antes e depois do HGP. Mostra um foco intenso em um pequeno número de genes codificadores de proteínas “superestrelas”, potencialmente em detrimento de um trabalho interessante que poderia ser feito em outros. Tem havido um pivô em direção a seções não codificadoras de proteínas do genoma e à compreensão das interações entre o material genético e as proteínas. E a descoberta de drogas foi baseada em apenas alguns alvos proteicos.
Algumas dessas tendências são familiares aos biólogos, mas quantificá-las e visualizá-las é considerá-las novamente.
Não há mundo sem um HGP para comparação. Portanto, é impossível dizer se essas tendências teriam surgido de qualquer maneira. Outros fatores, de maior poder de computação a métodos sofisticados de sequenciamento, também tiveram um papel em muitos desses desenvolvimentos. No entanto, é claro que o catálogo do HGP catalisou a revolução genética contínua.
Genes superstar
A percepção popular é que o HGP marcou o início de uma intensa busca por genes que codificam proteínas. Na verdade, o rascunho de papel do HGP de 2001 sinalizou o fim de uma caçada de décadas. De fato, a evidência do primeiro gene codificador de proteína surgiu em 1902, com a descoberta do hormônio secretina (gene SCT), 50 anos antes que a estrutura do DNA fosse descoberta e 75 anos antes que o sequenciamento do genoma se tornasse comum. Nossa análise mostra que, entre o início do HGP em 1990 e sua conclusão em 2003 (depois que o rascunho foi publicado em 2001), o número de genes humanos descobertos (ou “anotados”) cresceu drasticamente. Ele se estabilizou repentinamente em meados da década de 2000 em cerca de 20.000 genes codificadores de proteínas (consulte ‘Vinte anos de lixo, estrelas e drogas: elementos não codificantes’), muito aquém da estimativa forte de 100.000 previamente adotada por muitos na área científica comunidade.
Embora as descobertas de genes codificadores de proteínas tenham atingido um platô, o interesse em genes individuais cresceu rapidamente após o HGP. Todos os anos, desde 2001, entre 10.000 e 20.000 artigos mencionando genes codificadores de proteínas foram publicados (ver SI; Fig. S3).
No entanto, esse interesse se concentrou principalmente em apenas alguns genes. Antes de 1990, o HBA1 era o mais estudado por codificar uma das proteínas da hemoglobina adulta. A partir de 1990, a atenção se voltou para o CD4 (com base no número cumulativo de publicações), dado o envolvimento da proteína na imunidade das células T e como o receptor celular para o HIV. No entanto, o interesse por esses dois genes empalidece perto da explosão de atenção sobre os genes individuais após o esboço da seqüência de 2001 do HGP. Alguns genes de superstar, incluindo TP53, TNF e EGFR, tornaram-se o assunto de centenas de publicações por ano, com a maioria dos outros genes recebendo pouca atenção (consulte ?Impacto profundo? e ?Vinte anos de lixo, estrelas e drogas: genes de estrelas?). Descobrimos que, em 2017, 22% das publicações relacionadas a genes referiam-se a apenas 1% dos genes.

O estudo intenso é, naturalmente, justificado para genes que têm profunda importância biológica. Um bom exemplo é o TP53 – é crucial para o crescimento e morte celular, e leva ao câncer quando inativado ou alterado. Variações neste gene são encontradas em mais de 50% das sequências tumorais. É mencionado em 9.232 publicações entre 1976 e 2017 (ver SI, Fig. S4).
Pode-se supor que quanto mais se sabe sobre os mesmos genes, maior será o incentivo para explorar o resto do genoma. Em vez disso, o oposto aconteceu durante as últimas duas décadas: mais atenção foi dada a alguns poucos selecionados. Apesar de ser sinalizado como um problema potencial no décimo aniversário da publicação do projeto do genoma, não houve correção de curso.
Nosso trabalho anterior em outros sistemas muito diferentes, desde redes sociais humanas até a World Wide Web, indica que esse vasto desequilíbrio pode ser explicado por uma dinâmica “rico fica mais rico” enraizada em fatores sociais. É provável que, à medida que o número de artigos focados no TP53 aumenta, mais fácil é garantir financiamento, orientação, ferramentas e citações para trabalhos futuros no TP53, porque é uma aposta segura (ver SI; Figura S4). Na ciência das redes, esse fenômeno é denominado apego preferencial7. Na verdade, descobrimos que o número de novas publicações anuais com foco em um determinado gene é linearmente proporcional ao tamanho da literatura anterior sobre ele (ver SI, Fig. S6).
Um desafio agora para a biologia é separar as motivações para o que será estudado a seguir. Os pesquisadores estão investindo dinheiro, tempo e esforço no que é mais importante ou urgente, ou em mais do mesmo, porque isso certamente ganhará concessões e aplausos?
Não é lixo
Um grande debate antecedeu o início do HGP: valia a pena mapear as vastas regiões não codificantes do genoma que eram chamadas de DNA-lixo, ou a matéria escura do genoma? Graças em grande parte ao HGP, agora é reconhecido que a maioria das sequências funcionais no genoma humano não codifica proteínas. Em vez disso, elementos como longos RNAs não codificantes, promotores, potenciadores e incontáveis motivos reguladores de genes trabalham juntos para dar vida ao genoma. A variação nessas regiões não altera as proteínas, mas pode perturbar as redes que governam a expressão das proteínas.
Com o projeto de HGP em mãos, a descoberta de elementos não codificadores de proteínas explodiu. Até agora, esse crescimento ultrapassou a descoberta de genes codificadores de proteínas por um fator de cinco e não mostra sinais de desaceleração. Da mesma forma, o número de publicações sobre tais elementos também cresceu no período coberto por nosso conjunto de dados (1900 a 2017; ver SI, Fig. S3a). Por exemplo, existem milhares de artigos sobre RNAs não codificantes, que regulam a expressão gênica.
O HGP também ofereceu uma maneira de catalogar a variação genética humana, incluindo a dos SNPs. Outros grandes esforços reduziram o custo de criar perfis de diferenças comuns entre milhares de indivíduos; estes incluíram o International HapMap Project (cuja terceira e última fase foi concluída em 2010) e o 1000 Genomes Project (concluído em 2015). Esses conjuntos de dados, combinados com os avanços na análise estatística, deram início a estudos de associação do genoma (GWAS) de inúmeras características, incluindo altura, obesidade e suscetibilidade a doenças complexas como a esquizofrenia.
Existem agora mais de 30.000 artigos por ano ligando SNPs e traits. Uma grande fração dessas associações está nas regiões não codificantes antes descartadas (consulte SI, Tabela S3).
A função celular depende de ligações fracas e fortes entre o material genético e as proteínas. O mapeamento dessa rede agora complementa a perspectiva Mendeliana. Hoje, mais de 300.000 interações de rede regulatória foram mapeadas – proteínas se ligando a regiões não codificantes ou com outras proteínas.
Descoberta de drogas
Antes da década de 1980, as drogas eram encontradas em grande parte por acaso. Seus alvos moleculares e protéicos eram geralmente desconhecidos. Até 2001, a probabilidade de saber todos os alvos proteicos de um medicamento era inferior a 50% em qualquer ano. O HGP mudou isso. Agora, os alvos são conhecidos por quase todas as drogas licenciadas nos Estados Unidos a cada ano (consulte “Vinte anos de lixo, estrelas e drogas: alvos de drogas”).
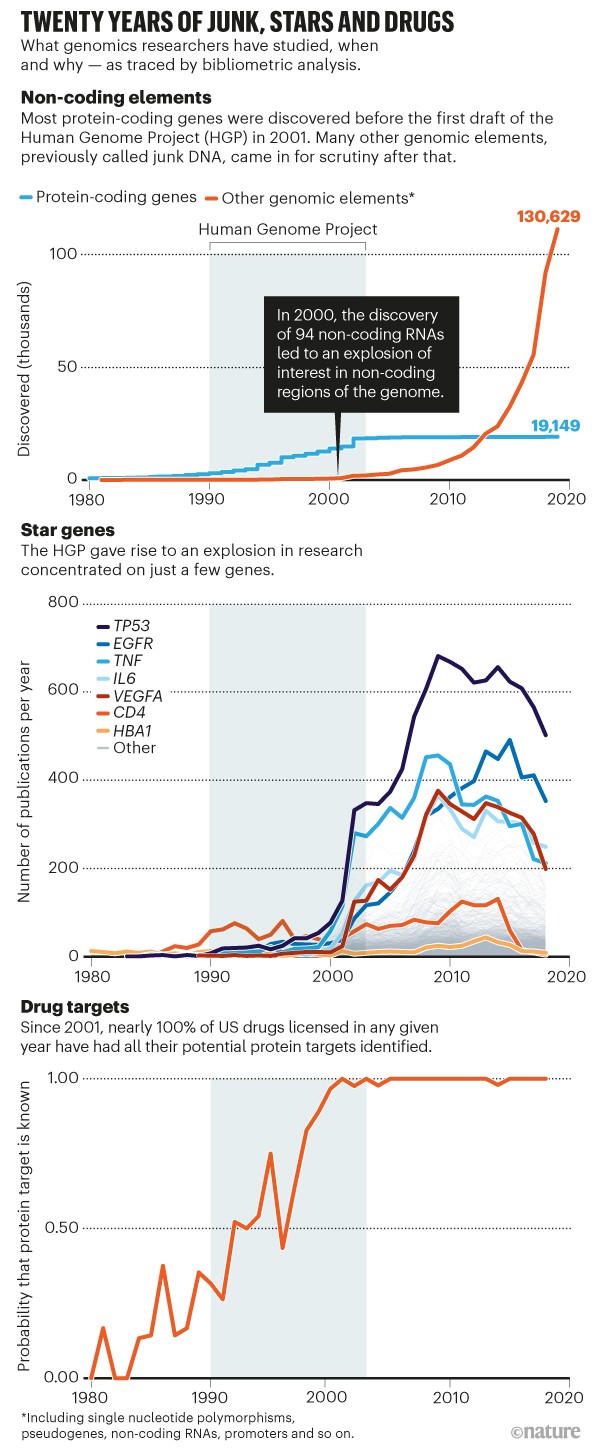
Das cerca de 20.000 proteínas reveladas pelo HGP como potenciais alvos de drogas, mostramos que apenas cerca de 10% – 2.149 – foram até agora alvo de drogas aprovadas (ver SI, Tabela S4 e Fig. S1). Isso deixa 90% do proteoma intocado pela farmacologia. Drogas experimentais em nosso conjunto de dados aumentam esse número para 3.119 (SI, Fig. S2). Novamente, a atenção dada a eles é altamente desigual. Cinco por cento de todos os medicamentos aprovados atualmente aprovados (99 moléculas distintas) têm como alvo a proteína ADRA1A, que está envolvida no crescimento e proliferação celular.
Como anteriormente, pode haver bons motivos para essa distorção. Algumas proteínas podem ser mais importantes para a saúde humana ou mais propensas a atuar como alvos de drogas. Alguns podem não ser drogáveis. Ou pode ser que existam muito mais proteínas que valem a pena explorar como alvos de drogas, se apenas os pesquisadores, financiadores e editores fossem menos avessos ao risco.
Dito isso, a maioria dos medicamentos de sucesso não tem como alvo direto os genes de doenças individuais. Em vez disso, eles têm como alvo as proteínas a uma ou duas interações de distância, modulando as consequências dos componentes defeituosos. Por exemplo, triagens em grande escala de drogas existentes que poderiam ser reaproveitadas para uso contra COVID-19 descobriram que apenas 1% dos candidatos promissores tinham como alvo uma proteína viral – a maioria eram drogas que modulavam proteínas humanas não diretamente envolvidas no SARS-CoV-2 atividade15. Essas drogas de rede têm um potencial enorme.
Rede vislumbrada
Em resumo, pensamos que o HGP é mais notável pela nova era da genômica que inaugurou do que pelo próprio catálogo de proteínas. Como mostra a teoria dos sistemas complexos, um levantamento preciso dos componentes é necessário – mas não suficiente – para entender qualquer sistema. A complexidade surge da diversidade das interações entre os componentes. Após 20 anos de pesquisas sobre o HGP, os biólogos agora têm um vislumbre da estrutura e dinâmica da rede que define a vida.
Publicado em 14/02/2021 07h59
Artigo original:
Estudo original:
Artigo relacionado:


