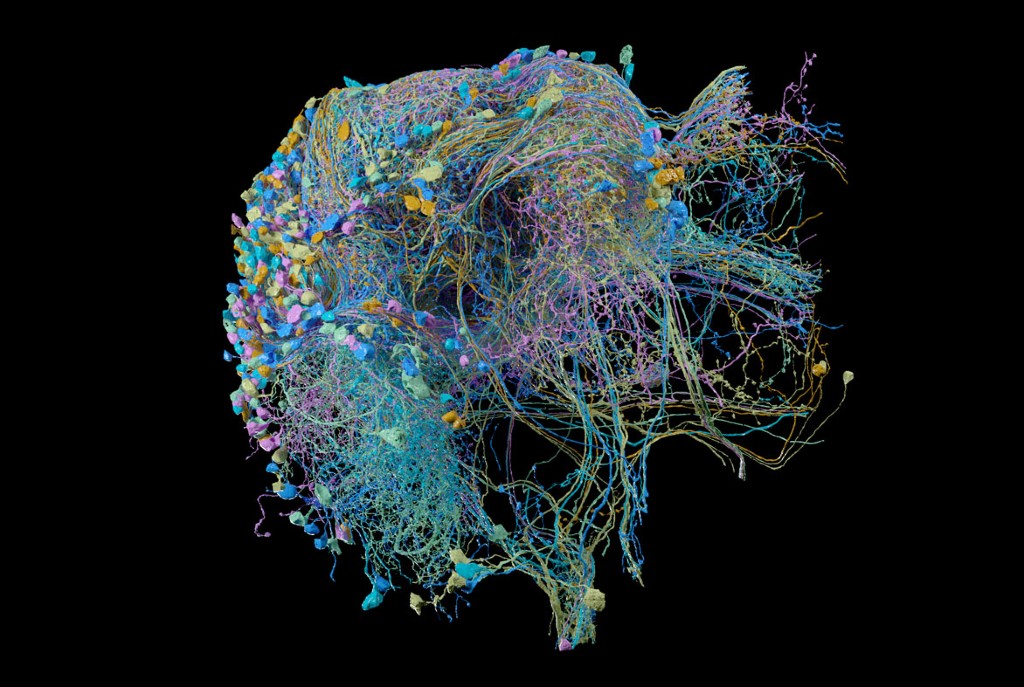
A inteligência artificial e a microscopia aprimorada tornam viável mapear o sistema nervoso em resoluções cada vez mais altas.
Existem 70 milhões de neurônios no cérebro do rato, e Moritz Helmstaedter deseja mapear todos eles. Ele era estudante de medicina na Universidade de Heidelberg, na Alemanha, quando psiquiatras sugeriram que alguns aspectos da psique humana carecem de explicação biológica. “Fiquei totalmente chocado”, lembra Helmstaedter, que agora é diretor do Instituto Max Planck para Pesquisa do Cérebro em Frankfurt, Alemanha.
Embora o cérebro permaneça um mistério, Helmstaedter estava convencido de que o que acontece lá “deve ser um fenômeno mecanicista no final, por mais complexo que seja”. Ele dedicou as últimas duas décadas a trabalhar esses mecanismos – e ele e outros neurocientistas estão finalmente começando a arranhar a superfície, um micrômetro cúbico de cada vez.
Começando na década de 1970, levou mais de uma década para desvendar o circuito neural do verme de um milímetro, Caenorhabditis elegans. Sondando a relação entre genes e comportamento, o biólogo Sydney Brenner e seus colegas do Laboratório MRC de Biologia Molecular em Cambridge, Reino Unido, traçaram laboriosamente os ramos finos e as conexões sinápticas de cada célula nervosa, codificando-as por cores manualmente em milhares de elétrons. impressões de micrografia. Esse mapa de fiação – o primeiro e único conjunto completo de conexões sinápticas no sistema nervoso de um animal – foi armazenado em um computador do tamanho de uma sala e publicado como o primeiro “conectoma” animal completo em um opus de 340 páginas em 1986.
Caenorhabditis elegans tem menos de 400 neurônios; cérebros humanos têm 86 bilhões. Portanto, por enquanto, os cientistas estão de olho em um marco intermediário: mapear o circuito neural de escala fina do mouse.
Mesmo com cerca de 1.000 vezes menos células, o cérebro do rato representa um desafio formidável, diz Jeff Lichtman, neurocientista da Universidade de Harvard em Cambridge, Massachusetts, que é um dos líderes de um consórcio global que visa reconstruir a fiação neural de um cérebro de rato durante a próxima década. “Estamos lidando com um conjunto de dados que estará na escala de um exabyte.” Um exabyte é um bilhão de gigabytes; todo o genoma humano pode ser representado em cerca de 1,5 gigabytes. Em termos de tamanho de dados, mapear o conectoma do cérebro do rato será “enorme em comparação com qualquer coisa que tenha sido feita como um único projeto”, diz ele. “Conectomas são simplesmente magnificamente complicados.”
No entanto, a tecnologia para tornar tal empreendimento possível está quase lá. Com os avanços na microscopia e inteligência artificial (IA), e a ajuda coletiva de jogadores humanos, os pesquisadores estão começando a mapear as redes neurais e suas conexões em resolução e escala cada vez maiores. Nos últimos anos, pequenos pedaços do cérebro, incluindo pedaços da retina e do córtex cerebral dos mamíferos, entraram em foco. E em setembro, pesquisadores que trabalham com as moscas-das-frutas Drosophila relataram a maior reconstrução até agora: 25.000 neurônios no hemibrain, um cubo de tecido medindo 250 micrômetros de lado e representando 40% do cérebro da mosca.
Estes não são meros exercícios de biologia grande. À medida que a conectômica empurra os limites tecnológicos e computacionais, os pesquisadores esperam explorar esses conjuntos de dados para aprender como as experiências são armazenadas no cérebro, com potenciais insights sobre autismo, esquizofrenia e outras “conectopatias”.
Desenvolvimentos iniciais
Depois que o diagrama de fiação neural de C. elegans lançou a conectômica em 1986, o campo ficou em silêncio, diz Helmstaedter. Era uma questão de tecnologia: os pesquisadores não tinham como, além do que a equipe de Brenner havia feito, sondar circuitos neurais em escalas de conectoma.
Como estudante de doutorado no início dos anos 2000, Helmstaedter colocou eletrodos nas células nervosas para descobrir quais estavam conectadas eletricamente, uma abordagem que poderia permitir o registro simultâneo de quatro ou cinco neurônios. No entanto, as redes têm centenas ou milhares de células nervosas e milhões de conexões. “Para realmente mapear os circuitos, precisávamos de algo mais”, diz ele.
Isso aconteceu em 2004. Winfried Denk, então no Instituto Max Planck de Pesquisa Médica em Heidelberg, e seus colegas instalaram uma ferramenta de corte de precisão chamada micrótomo na câmara de vácuo de um microscópio eletrônico (EM), tornando possível automatizar a nanoescala imagem. Revitalizou o campo.
Chamado de microscopia eletrônica de varredura em bloco serial (SBEM), o método de Denk envolve o carregamento de um bloco de tecido na máquina, que então imagina automaticamente o rosto exposto, raspa a camada superior do tecido e se repete por dias ou semanas. Em 2013, a equipe de Denk, liderada por Helmstaedter, um ex-pós-doutorado no laboratório, usou o SBEM para mapear um conjunto completo de conexões sinápticas para 950 neurônios na retina do rato. Este foi um empreendimento significativo: o custo, incluindo equipamento, salários e cerca de $ 300.000 (US $ 350.000) em taxas pagas a estudantes de graduação para rastrear circuitos em todos os conjuntos de dados EM, totalizou cerca de $ 2 milhões. E revelou novos subtipos de células. Mas, além disso, o trabalho forneceu um mapa abrangente para os pesquisadores identificarem parceiros de interação para células de interesse, diz Helmstaedter – “como usar um mapa de ruas para navegação versus tentativa e erro”.
Worm matemática
Os alunos que traçaram esses circuitos neurais o fizeram usando computadores. Essa mudança começou no início de 2000, quando os pesquisadores começaram a adotar uma abordagem computacional para mapear o conectoma. Isso não era aprendizado de máquina; os humanos ainda faziam o trabalho. Mas em vez de traçar neurônios no papel com lápis de cor, como a equipe de Brenner fez, eles clicaram com o mouse em pilhas de imagens digitalizadas.
O biólogo Scott Emmons do Albert Einstein College of Medicine em Nova York e sua equipe, por exemplo, digitalizaram as imagens originais de Brenner e usaram uma abordagem computacional para mapear os circuitos que regulam os comportamentos de acasalamento na cauda de um C. elegans macho. (O esforço de 1986 se concentrou no outro sexo de C. elegans, o hermafrodita.)

Emmons então contratou um estatístico para aplicar a teoria dos grafos – um ramo da matemática usado para analisar redes como rotas de ônibus ou mapas da propagação de doenças – para calcular a força das conexões sinápticas e descobrir quais partes da rede conectoma conduzem comportamentos específicos. Embora os matemáticos tenham estudado redes neurais artificiais por décadas, o trabalho de C. elegans, relatado em 2012, foi considerado a primeira análise quantitativa de uma rede neural natural, diz Emmons.
Estendendo esses esforços, em 2019 Emmons e seus colegas publicaram 7 conectomas quantitativos dos sistemas nervosos completos de ambos os sexos de C. elegans, compreendendo 687 neurônios. Um dos objetivos era determinar como a conectividade diferia entre o homem e o hermafrodita. Mas distinguir as diferenças baseadas no sexo da variabilidade biológica natural entre os indivíduos provou ser um osso duro de roer, atrasando o trabalho por um ano inteiro e exigindo ainda mais matemática. No final, a equipe concluiu que até 30% das conexões neurais podem refletir diferenças reais com base no sexo, diz Emmons, em oposição às disparidades decorrentes de condições de criação ou erros metodológicos.
Complexidade inesperada
Rastrear o circuito neural através do cérebro de um minúsculo verme requer milhares de seções de 30 a 50 nanômetros. “Mesmo se você for um deus, você não pode cortar 5.000 seções sem bagunçar nada”, diz Mei Zhen, neurocientista da Universidade de Toronto, no Canadá.
Agora tente fazer isso oito vezes. Em uma análise descrita em uma pré-impressão bioRxiv em maio de 8, pesquisadores liderados por Zhen, Lichtman e o físico de Harvard Aravinthan Samuel reconstruíram conectomas em oito estágios de desenvolvimento, para aprender como o diagrama de fiação de C. elegans muda conforme o verme amadurece da larva inicial ao adulto. Eles seccionaram a maioria de suas amostras com um ultramicrótomo de coleta de fita automatizado, desenvolvido no laboratório de Lichtman, que coleta seções seriais e as coloca em sequência em uma bobina para imagens subsequentes por um microscópio eletrônico de varredura. Ele “transforma um verme em um rolo de fita”, diz Zhen.
Usando-o, a equipe detectou enormes diferenças na fiação dos vermes. Mesmo entre animais geneticamente idênticos, diz Zhen, cerca de 43% das conexões não eram as mesmas.
No Laboratório de Biologia Molecular do MRC, a neurocientista de sistemas Marta Zlatic estuda outra criatura semelhante a um verme: a larva de Drosophila. Medindo de 4 a 8 milímetros de comprimento, as larvas são maiores do que C. elegans, mas têm 10 a 20 vezes menos neurônios cerebrais do que as moscas adultas. “Portanto, podemos reconstruir circuitos muito mais rápido e podemos fazer isso em muitos indivíduos”, diz Zlatic, que combina a conectômica com outras técnicas para correlacionar a estrutura neural com a função.
Em um estudo publicado em março, a equipe de Zlatic analisou 102 pares de neurônios envolvidos na prevenção de odores. Eles descobriram que mesmo nesses minúsculos insetos, redes celulares distantes regulam o aprendizado. “Quando um animal aprende algo novo sobre um odor, com que rapidez e quão bem ele aprende isso será influenciado por tudo o mais que ele já aprendeu sobre aquele odor”, diz ela.
Aprendizado de máquina
Ampliar essas análises para animais como Drosophila adulta ou camundongos, que são capazes de aprendizado de ordem superior, é muito mais desafiador. Mas aí também está havendo progresso.
Em um estudo de 2019, Helmstaedter e seus colegas usaram o SBEM para criar imagens e reconstruir uma partícula do cérebro de um rato em uma região que processa a entrada sensorial. Medindo 500.000 micrômetros cúbicos, o volume continha cerca de 2,7 metros de fiação neuronal e 400.000 conexões sinápticas. Ele continha apenas 89 neurônios, mas era 300 vezes maior do que as reconstruções anteriores do córtex cerebral de mamíferos.
A reconstrução desses circuitos com as técnicas padrão da época – passando de corte em corte, rastreando manualmente cada célula nervosa – levaria centenas de milhares de horas, estima Helmstaedter. Portanto, sua equipe combinou algoritmos de processamento de imagem automatizado com abordagens de IA de aprendizado de máquina e concentrou o esforço humano na marcação de ramos de neurônios, permitindo que os computadores tratassem da reconstrução volumétrica. Isso reduziu a carga de trabalho para 20.000 horas – ainda o equivalente a 10 pessoas trabalhando em tempo integral por um ano. Outras melhorias na IA aceleraram ainda mais o processo, treinando computadores para avaliar as reconstruções montadas nas máquinas e solicitando ajuda humana apenas quando necessário.
Enquanto isso, pesquisadores do Campus de Pesquisa Janelia do Howard Hughes Medical Institute (HHMI) em Ashburn, Virginia, voltaram seus olhos para a Drosophila. O cérebro de uma mosca da fruta tem muito menos neurônios do que o cérebro de um rato, mas seus fios são mais finos e mais densos, diz Shan Xu, um físico aplicado da Janelia. Rastrear essas conexões até a conclusão exigiria resolução de 8 nm – 3-6 vezes o que era possível com a varredura EM quando Xu se juntou ao HHMI em 2009. Dada a tecnologia disponível então, diz ele, a imagem sozinha teria levado uma década.
E esse não foi nem o maior desafio. Para rastrear cada processo neuronal nesta resolução, as imagens tinham que ser perfeitas; uma lacuna de 100 nm pode tornar o conjunto de dados inútil. “Você gostaria de operar uma máquina por uma década sem erros”, diz Xu.
Xu passou vários anos investigando meticulosamente os modos de falha e as condições que os desencadearam. “Antes de falhar, quais são os sinais reveladores? Quais são os indicadores que posso capturar e interromper imediatamente a operação? E então como posso reiniciar a partir daí sem problemas?” ele perguntou. Em vez da perfeição, ele optou pela inteligência, construindo um sistema que poderia se desligar quando uma falha do computador, falha da bomba de vácuo ou outro mau funcionamento se avizinhava. Essa mudança de mentalidade “mudou completamente a imagem”, diz Xu. Isso ajudou sua equipe a refinar outra tecnologia de face de bloco, FIB-SEM, que corta usando um feixe de íons focalizado em vez de um micrótomo e produz imagens de alta resolução com poucos defeitos.
Xu e seus colegas executaram dois desses sistemas FIB-SEM em paralelo por cerca de dois anos. Então, depois que um computador alinhou as pilhas de imagens EM, uma equipe de 50 funcionários de HHMI trabalhou em tempo integral por um ano para revisá-las. Analisando milhares de imagens por dia, os revisores aprenderam rapidamente a detectar erros – por exemplo, segmentos unidos incorretamente ou peças órfãs que precisavam ser conectadas. “Você desenvolve seu olho para o tipo de neurônio que está olhando”, diz Erika Neace, uma desenvolvedora de análises e revisora veterana da Janelia. Ao todo, a equipe rastreou 25.000 neurônios e suas 20 milhões de conexões no hemibrain3.
Separadamente, outra equipe da Janelia fotografou o cérebro da mosca usando um microscópio eletrônico de transmissão (TEM), uma tecnologia mais antiga que passa elétrons através de espécimes finos em vez de escanear uma superfície como o FIB-SEM faz. Ajustando o microscópio com uma série de câmeras e componentes robóticos para processar espécimes maiores em velocidades mais altas, eles fotografaram um cérebro de Drosophila adulto inteiro. Eles então validaram o conjunto de dados resultante – todos os 106 terabytes dele – rastreando meticulosamente os circuitos no corpo do cogumelo, uma estrutura cerebral importante para o aprendizado e a memória.
Hoje, os pesquisadores estão terceirizando o processo de anotação, criando jogos online como bancos de teste para conectômica em escala precisa usando IA e voluntários humanos. Em agosto, pesquisadores liderados pelos neurocientistas Sebastian Seung e Mala Murthy da Universidade de Princeton em New Jersey lançaram uma plataforma semelhante a um jogo chamada FlyWire, que apresenta pedaços de neurônios identificados por IA do conjunto de dados de cérebro de mosca de Janelia para os jogadores montarem. Com base no Eyewire – um jogo anterior desenvolvido pelo laboratório Seung usando um conjunto menor de imagens de retina do mouse – o FlyWire complementa a plataforma de visualização Neuroglancer do Google com ferramentas que permitem que vários usuários visualizem e editem neurônios no mesmo conjunto de dados ao mesmo tempo.
Ao contrário da reconstrução do hemibrain de Xu, que foi processada por uma equipe interna de revisão, o FlyWire convida a contribuição de todos. E essas contribuições melhoram o sistema com o tempo, diz Seung. Quando um usuário edita um neurônio clicando em um botão para dividir ou mesclar peças, essa ação envia uma mensagem que treina a máquina para detectar reconstruções erradas e, com o tempo, corrigi-las. “Este é um momento realmente profundo e filosoficamente interessante”, diz Lichtman. “As máquinas estão aprendendo a ser mais inteligentes ao estudar a fiação de máquinas que são fundamentalmente mais inteligentes – máquinas biológicas.”
Seung agora está colaborando com pesquisadores da Baylor College of Medicine em Houston, Texas, e do Allen Institute for Brain Science em Seattle, Washington, para construir uma nova comunidade online, Pyr (como em, ‘Pyr no cérebro’), para mapear um pedaço ainda maior do cérebro do rato. Em outubro, a equipa do Allen Institute, liderada por Clay Reid e Nuno da Costa, descreveu o método utilizado para a recolha desses dados. Usando seis TEMs da década de 1980 adaptados com câmeras de grande campo de visão e um sistema de manuseio de amostra de bobina a bobina personalizado, a equipe obteve imagens de um milímetro cúbico do córtex visual do mouse em 6 meses, produzindo 2 petabytes de dados. Agora, a equipe está tentando dimensionar suas operações para o nível do cérebro inteiro, diz Wenjing Yin, um cientista do Allen Institute que ajudou a desenvolver o canal de imagem. “Há muitas perguntas e problemas que precisamos resolver”, diz ela.
Com cerca de 70 milhões de neurônios de rato ainda não mapeados, a comunidade conectoma terá seu trabalho interrompido. Mas Helmstaedter está otimista. “É um momento extremamente emocionante para mapear um novo território”, diz ele, “com todas as surpresas que isso sempre traz”.
Publicado em 24/10/2020 01h21
Artigo original:
Estudo original:
Achou importante? Compartilhe!
Assine nossa newsletter e fique informado sobre Astrofísica, Biofísica, Geofísica e outras áreas. Preencha seu e-mail no espaço abaixo e clique em “OK”:




