
Cientistas do La Jolla Institute for Immunology (LJI) descobriram como a perda de enzimas TET pode levar ao linfoma de células B. Sua pesquisa, publicada na Nature Immunology, poderia potencialmente abrir oportunidades para projetar estratégias de tratamento de drogas para atingir células malignas em muitos tipos de câncer.
A nova pesquisa foi liderada pela professora LJI Anjana Rao, Ph.D., no Centro LJI de Imunoterapia do Câncer, e os experimentos foram liderados pelo instrutor LJI Vipul Shukla, Ph.D., (em breve será professor assistente na Northwestern University) e a estudante de pós-graduação da UC San Diego Daniela Samaniego-Castruita.
A nova pesquisa ajuda os cientistas a finalmente vincular dois fenômenos perigosos nas células cancerígenas.
Em estudos anteriores, os cientistas detectaram mutações que fazem com que as enzimas TET percam sua função em muitos pacientes com câncer no sangue e câncer sólido. Os pesquisadores também descobriram que a instabilidade genômica, como quebras de fita dupla no código do DNA, são uma característica comum em células cancerígenas.
Neste projeto, os cientistas exploraram uma maneira potencial pela qual a deficiência de TET está ligada à instabilidade genômica.
“Este estudo fornece insights sobre uma questão importante no campo”, diz Shukla.
Ao estudar um modelo de linfoma em camundongo, os pesquisadores descobriram que a exclusão das enzimas TET2 e TET3 em células B maduras teve enormes consequências para a homeostase das células B. “Os camundongos com deficiência de TET desenvolveram linfoma e observamos um aumento nas marcas associadas à instabilidade genômica, como quebras de fita dupla”, diz Samaniego-Castruita.
A equipe então realizou análises genômicas em busca de pistas sobre o que estava acontecendo no nível molecular. Eles viram que sem TET2 e TET3, o DNA ficou repleto de estruturas de DNA incomuns chamadas G-quadruplexes e R-loops.
O DNA normalmente tem duas fitas paralelas uma à outra, como dois trilhos de uma escada. R-loops aparecem quando um terceiro trilho, feito de RNA, desliza e força uma lacuna entre os dois trilhos de DNA. G-quadruplexes agem como nós nos trilhos de DNA. Tanto R-loops quanto G-quadruplexes tornam difícil para os dois trilhos originais de DNA “descompactarem” enquanto a célula tenta ler o código de DNA e manter a célula funcionando corretamente.
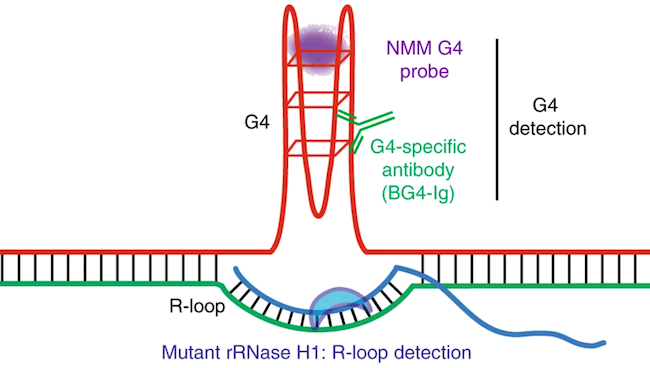
Shulka e Samaniego-Castruita examinaram essas estruturas de DNA em profundidade graças ao financiamento do Tullie and Rickey Families SPARK Awards for Innovations in Immunology do La Jolla Institute. “Essas estruturas representam locais no DNA que são muito mais frágeis do que outras regiões”, diz Shukla. “Com este estudo, descobrimos que as enzimas TET talvez estejam relacionadas à regulação dessas estruturas, o que poderia explicar um mecanismo de aquisição de instabilidade genômica na ausência de enzimas TET”.
Quando se trata de malignidades de células B, G-quadruplexes e R-loops parecem ser um elo perdido entre mutações TET reveladoras e instabilidade genômica perigosa.
Então, se G-quadruplexes e R-loops estão causando problemas, existe uma maneira de impedi-los de se formar?
Shukla e Samaniego-Castruita observaram que DNMT1 foi regulado positivamente em células B deficientes em TET. DNMT1 é uma enzima chave responsável pela manutenção de marcas no DNA chamadas “metilação do DNA”. A metilação do DNA é uma importante marca regulatória no genoma e normalmente é removida pela atividade das enzimas TET.
Sem as enzimas TET, a troca normal das marcas de metilação do DNA foi quebrada. Assim, em seu próximo experimento, os cientistas também deletou o gene Dnmt1 em células B deficientes em TET em camundongos para testar se os níveis de quadruplexes G e R-loops poderiam ser alterados após a remoção da proteína DNMT1.
De fato, a exclusão de DNMT1 foi associada a um atraso notável no desenvolvimento de linfomas de células B agressivos. A exclusão de DNMT1 também foi associada à diminuição dos níveis de G-quadruplexes e R-loops, diz Samaniego-Castruita.
Os pesquisadores enfatizam que a regulação de G-quadruplexes e R-loops pode ser apenas uma maneira pela qual as enzimas TET controlam a estabilidade genômica. Há também mais trabalho a ser feito para descobrir as etapas precisas que levam as células deficientes em TET a acumular essas estruturas enigmáticas no DNA. Algum dia, a equipe espera elaborar estratégias através das quais G-quadruplexes e R-loops possam ser direcionados para ajudar pacientes com câncer.
À medida que o Rao Lab continua investigando os efeitos das enzimas TET, Shukla se juntará ao corpo docente do Departamento de Biologia Celular e do Desenvolvimento da Northwestern University neste inverno. Ele planeja lançar seu próprio laboratório focado no estudo de conformações estruturais alternativas no DNA.
Publicado em 01/03/2022 17h26
Artigo original:
Estudo original:


