Em apenas oito anos, o CRISPR-Cas9 tornou-se o editor de genoma preferido para pesquisa básica e terapia genética. Mas o CRISPR-Cas9 também gerou outras ferramentas de manipulação de DNA potencialmente poderosas que poderiam ajudar a corrigir mutações genéticas responsáveis por doenças hereditárias.
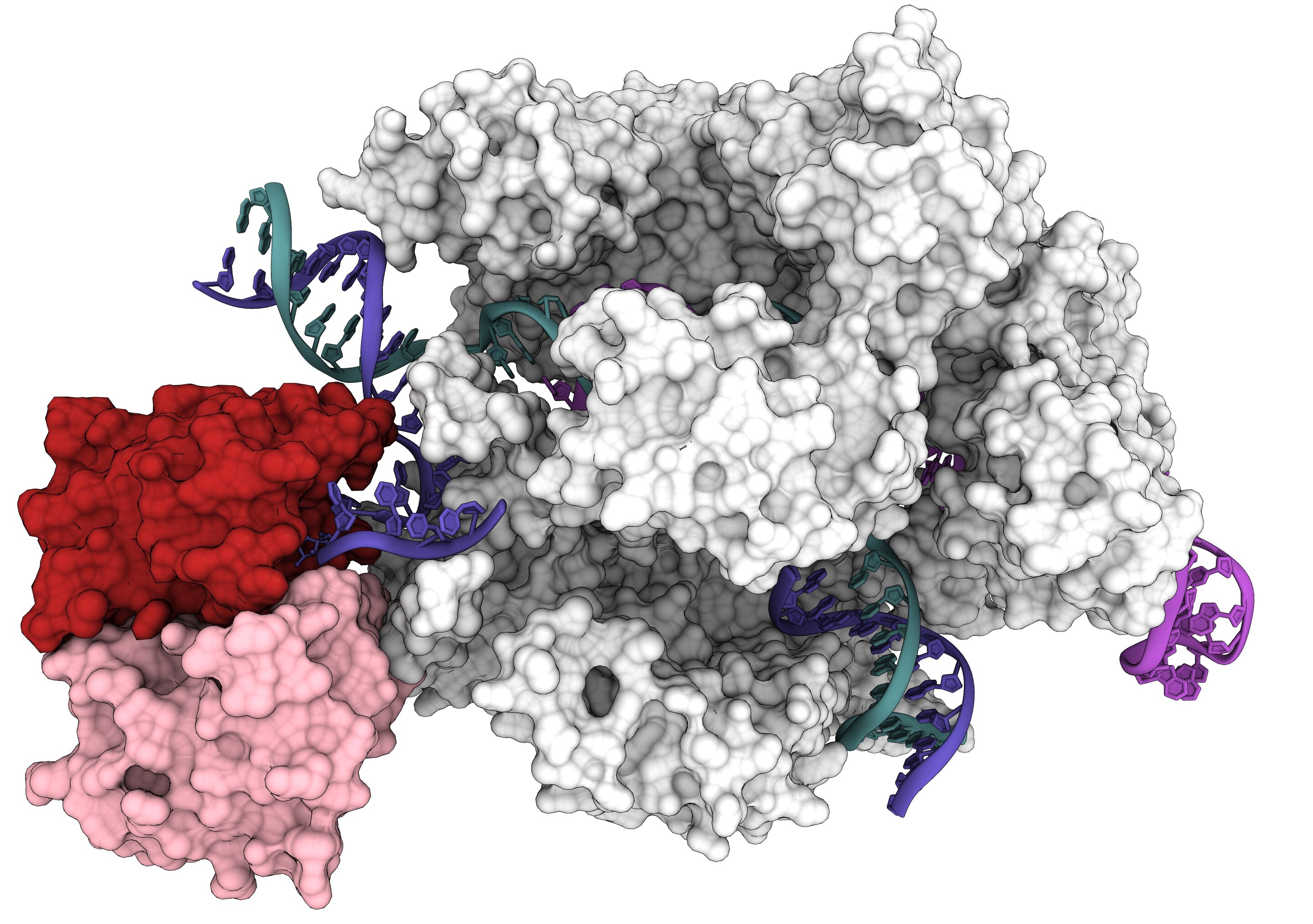
Pesquisadores da Universidade da Califórnia, Berkeley, agora obtiveram a primeira estrutura 3D de uma das ferramentas mais promissoras: editores de bases, que se ligam ao DNA e, em vez de cortar, substituem precisamente um nucleotídeo por outro.
Criados pela primeira vez há quatro anos, os editores de bases já estão sendo usados na tentativa de corrigir mutações de nucleotídeo único no genoma humano. Os editores de base agora disponíveis poderiam tratar de cerca de 60% de todas as doenças genéticas conhecidas – potencialmente mais de 15.000 doenças hereditárias – causadas por uma mutação em apenas um nucleotídeo.
A estrutura 3D detalhada, relatada na edição de 31 de julho da revista Science, fornece um roteiro para ajustar editores de base para torná-los mais versáteis e controláveis para uso em pacientes.
“Pudemos observar pela primeira vez um editor de base em ação”, disse Gavin Knott, pós-doutorado em UC Berkeley. “Agora podemos entender não apenas quando funciona e quando não funciona, mas também projetar a próxima geração de editores de base para torná-los ainda melhores e mais adequados clinicamente”.
Um editor de base é um tipo de proteína de fusão Cas9 que emprega um Cas9 parcialmente desativado – suas tesouras de corte são desativadas para cortar apenas uma fita do DNA – e uma enzima que, por exemplo, ativa ou silencia um gene ou modifica áreas adjacentes de DNA. Como o novo estudo relata a primeira estrutura de uma proteína de fusão Cas9, ele pode ajudar a orientar a invenção de inúmeras outras ferramentas de edição de genes baseadas em Cas9.
“Na verdade, vemos pela primeira vez que os editores de base se comportam como dois módulos independentes: você tem o módulo Cas9 que lhe dá especificidade e, em seguida, um módulo catalítico que fornece a atividade”, disse Audrone Lapinaite, ex-UC Berkeley pós-doutorado que agora é professor assistente na Arizona State University em Tempe. “As estruturas que obtivemos deste editor de base vinculadas ao seu alvo realmente nos permitem pensar nas proteínas de fusão Cas9, em geral, dando-nos ideias de qual região do Cas9 é mais benéfica para a fusão de outras proteínas”.
Lapinaite e Knott, que recentemente aceitaram uma posição como pesquisador na Universidade Monash, na Austrália, são co-primeiros autores do artigo.
Estrutura Cryo-EM do editor de base (ABE8e) no ato de editar o DNA. Crédito: Gavin J. Knott (PDB: 6VPC)
Editando uma base por vez
Em 2012, os pesquisadores primeiro mostraram como reengenharia de uma enzima bacteriana, Cas9, e transformá-la em uma ferramenta de edição de genes em todos os tipos de células, de bacteriana a humana. A ideia da bioquímica da UC Berkeley Jennifer Doudna e sua colega francesa Emmanuelle Charpentier, CRISPR-Cas9, transformou a pesquisa biológica e trouxe a terapia genética para a clínica pela primeira vez em décadas.
Os cientistas rapidamente cooptaram o Cas9 para produzir uma série de outras ferramentas. Basicamente, uma mistura de proteínas e RNA, o Cas9 tem como alvo preciso um trecho específico de DNA e, em seguida, o corta com precisão, como uma tesoura. A função de tesoura pode ser quebrada, no entanto, permitindo que Cas9 alveje e ligue o DNA sem cortar. Dessa forma, Cas9 pode transportar enzimas diferentes para regiões-alvo do DNA, permitindo que as enzimas manipulem genes.
Em 2016, David Liu, da Universidade de Harvard, combinou um Cas9 com outra proteína bacteriana para permitir a substituição cirurgicamente precisa de um nucleotídeo por outro: o primeiro editor de base.
Embora o editor de base da adenina inicial tenha sido lento, a versão mais recente, chamada ABE8e, é incrivelmente rápida: ela completa quase 100% das edições básicas pretendidas em 15 minutos. No entanto, o ABE8e pode ser mais propenso a editar pedaços de DNA não intencionais em um tubo de ensaio, criando potencialmente o que é conhecido como efeitos fora do alvo.
A estrutura recém-revelada foi obtida com uma técnica de imagem de alta potência chamada microscopia crioeletrônica (cryoEM). Os ensaios de atividade mostraram por que o ABE8e é propenso a criar mais edições fora do alvo: A proteína deaminase fundida ao Cas9 está sempre ativa. À medida que o Cas9 pula em torno do núcleo, ele se liga e libera centenas ou milhares de segmentos de DNA antes de encontrar o alvo pretendido. A desaminase anexada, como um canhão solto, não espera uma combinação perfeita e geralmente edita uma base antes que o Cas9 descanse em seu alvo final.
Saber como o domínio efetor e o Cas9 estão vinculados pode levar a um redesenho que torna a enzima ativa somente quando o Cas9 encontra seu alvo.
“Se você realmente deseja projetar proteínas de fusão verdadeiramente específicas, precisa encontrar uma maneira de tornar o domínio catalítico mais parte do Cas9, para que ele pareça quando o Cas9 está no alvo correto e só então é ativado, em vez de ser ativo o tempo todo “, disse Lapinaite.
A estrutura do ABE8e também identifica duas alterações específicas na proteína deaminase que a fazem funcionar mais rapidamente do que a versão inicial do editor de base, ABE7.10. Essas duas mutações pontuais permitem que a proteína agarre o DNA com mais força e substitua A com mais eficiência por G.
“Como biólogo estrutural, eu realmente quero olhar para uma molécula e pensar em maneiras de melhorá-la racionalmente. Essa estrutura e a bioquímica associada realmente nos dão esse poder”, acrescentou Knott. “Agora podemos fazer previsões racionais de como esse sistema se comportará em uma célula, porque podemos vê-lo e prever como ele vai quebrar ou prever maneiras de torná-lo melhor”.
Publicado em 01/08/2020 14h54
Artigo original:
Estudo original:
Achou importante? Compartilhe!
Assine nossa newsletter e fique informado sobre Astrofísica, Biofísica, Geofísica e outras áreas. Preencha seu e-mail no espaço abaixo e clique em “OK”:




