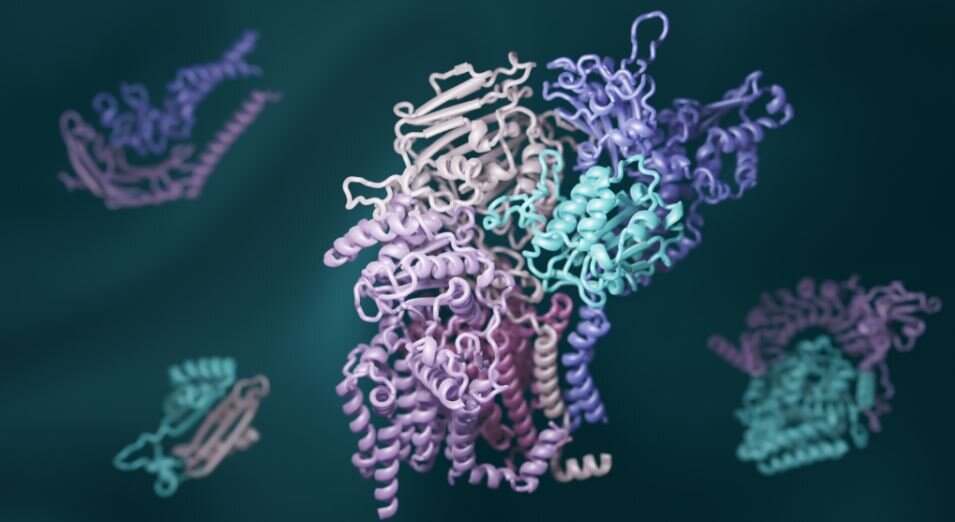
Os cientistas agora estão combinando avanços recentes em análise evolutiva e aprendizado profundo para construir modelos tridimensionais de como a maioria das proteínas em eucariotos interagem. (Eucariotos são organismos cujas células têm um núcleo ligado à membrana para conter materiais genéticos.)
O esforço de pesquisa tem implicações para a compreensão dos processos bioquímicos comuns a todos os animais, plantas e fungos. O trabalho de acesso aberto aparece em 11 de novembro na revista Science.
Como parte de uma colaboração multi-institucional, o laboratório de David Baker no UW Medicine Institute for Protein Design ajudou a guiar esse novo desenvolvimento.
“Para realmente entender as condições celulares que dão origem à saúde e às doenças, é essencial saber como diferentes proteínas em uma célula trabalham juntas”, disse Baker. “Neste artigo, fornecemos informações detalhadas sobre as interações de proteínas para quase todos os processos centrais em células eucarióticas. Isso inclui mais de uma centena de interações que nunca foram vistas antes.”
As proteínas, os burros de carga de todas as células, raramente agem sozinhas. Muitas vezes, diferentes proteínas precisam se encaixar para formar complexos precisos que realizam tarefas específicas. Isso pode incluir a leitura de genes, digestão de nutrientes e resposta a sinais de células vizinhas e do mundo exterior. Quando os complexos de proteínas funcionam mal, podem ocorrer doenças.
“Este trabalho mostra que o aprendizado profundo pode agora gerar percepções reais sobre questões de décadas atrás na biologia – não apenas como uma proteína em particular se parece, mas também quais proteínas se juntam para interagir”, disse o autor sênior Qian Cong, professor assistente de biofísica da o Centro Médico Southwestern da Universidade do Texas.
Para mapear exaustivamente as interações que dão origem aos complexos de proteínas, uma equipe de biólogos estruturais da UW Medicine, do Southwestern Medical Center da University of Texas, da Harvard University e de várias outras instituições examinou todas as sequências de genes conhecidas na levedura. Usando análises estatísticas avançadas, eles identificaram pares de genes que naturalmente adquirem mutações de forma interligada. Eles raciocinaram que essas mutações compartilhadas são um sinal de que as proteínas que os genes codificam devem interagir fisicamente.
Os pesquisadores também usaram um novo software de aprendizagem profunda para modelar as formas tridimensionais dessas proteínas em interação. RoseTTAFold, inventado na UW Medicine, e AlphaFold, inventado pela subsidiária da Alphabet DeepMind, foram usados para gerar centenas de imagens detalhadas de complexos de proteínas.
“À medida que os métodos de computador se tornam mais poderosos, é mais fácil do que nunca gerar grandes quantidades de dados científicos, mas para dar sentido a isso ainda são necessários especialistas científicos”, disse Baker, que é professor de bioquímica da Escola de Medicina da Universidade de Washington. “Então, recrutamos uma vila de biólogos especialistas para interpretar nossos modelos de proteína 3-D. Esta é a ciência da comunidade no seu melhor.”
As centenas de complexos de proteínas recentemente identificados fornecem informações valiosas sobre como as células funcionam. Por exemplo, um complexo contém a proteína RAD51, que é conhecida por desempenhar um papel fundamental no reparo do DNA e na progressão do câncer em humanos. Outra inclui a enzima mal compreendida glicosilfosfatidilinositol transamidase, que tem sido implicada em distúrbios do neurodesenvolvimento e câncer em humanos. Entender como essas e outras proteínas interagem pode abrir a porta para o desenvolvimento de novos medicamentos para uma ampla gama de distúrbios de saúde.
As estruturas de proteínas geradas neste trabalho estão disponíveis para download no ModelArchive. Os pesquisadores agradecem e lembram o falecido John Westbrook do Protein Data Bank por seu apoio no estabelecimento de formatos e código de software para permitir a deposição eficiente dos modelos no arquivo. O artigo da Science relatando os resultados estava em preparação no momento da morte de Westbrook.
O projeto sobre estruturas computadas de complexos de proteínas eucarióticas centrais foi liderado por Ian Humphrey, Aditya Krishnakumar e Minkyung Baek, todos da UW Medicine, bem como Jimin Pei do University of Texas Southwestern Medical Center. As instituições colaboradoras incluem UW Medicine, UT Southwestern, Harvard University, Wayne State University, Cornell University, MRC Laboratory of Molecular Biology, Memorial Sloan Kettering Cancer Center, Gerstner Sloan Kettering Escola de Graduação em Ciências Biomédicas, Fred Hutchinson Cancer Research Center, Columbia University, University de Würzburg, Hospital de Pesquisa Infantil St Jude, Instituto FIRC de Oncologia Molecular e Istituto di Genetica Molecolare, Consiglio Nazionale delle Ricerche.
Publicado em 24/11/2021 11h04
Artigo original:
Estudo original:
Artigo relacionado:


