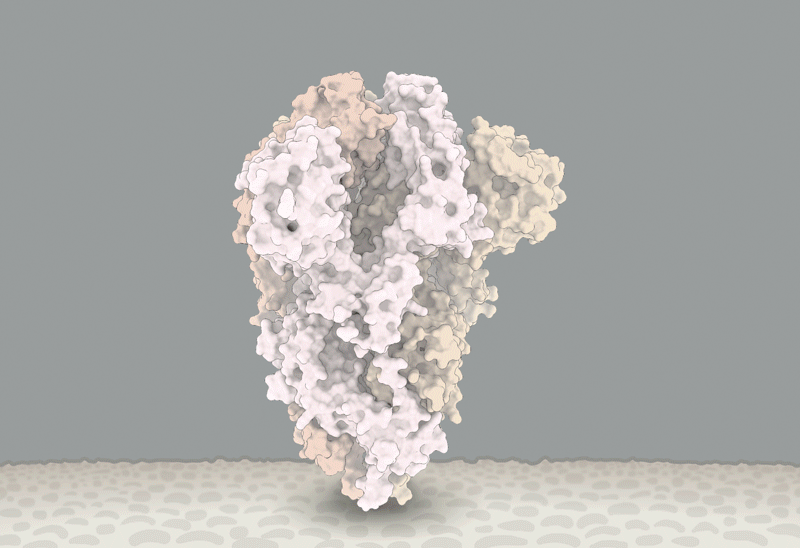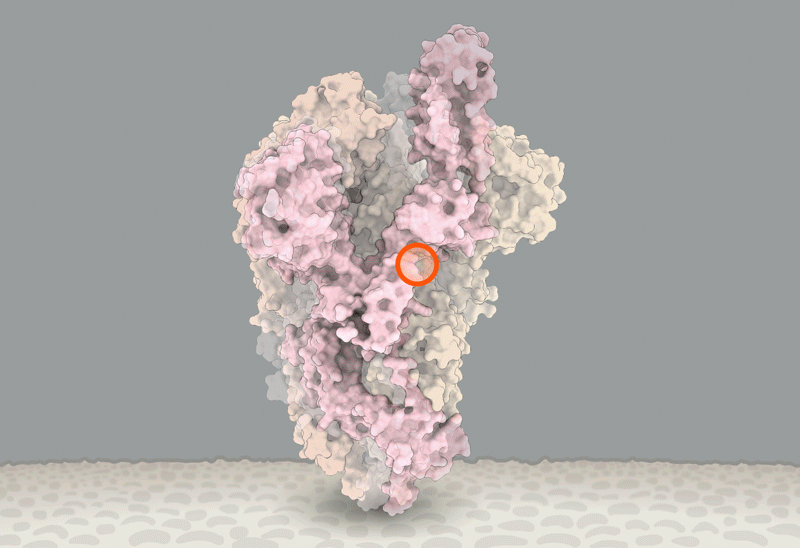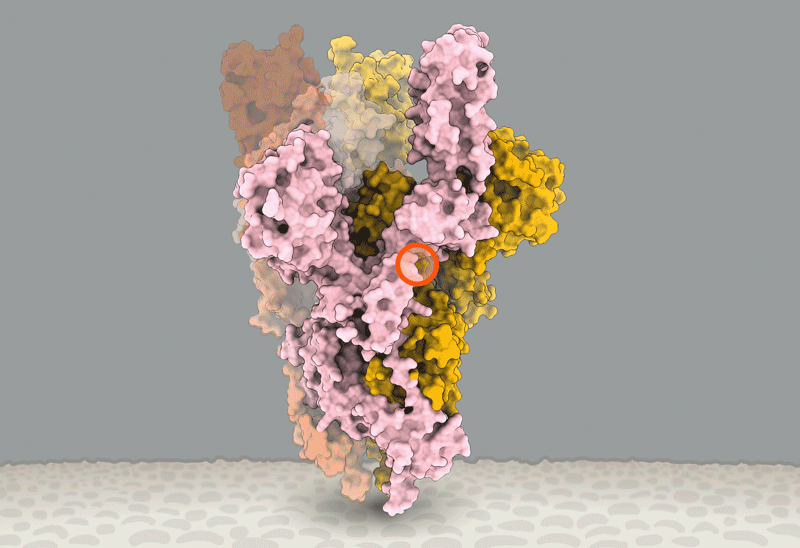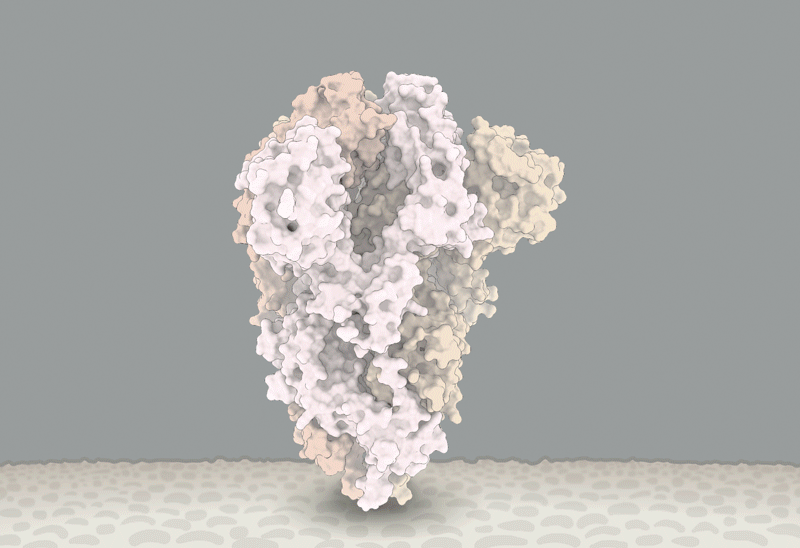
Diferentes cepas de SARS-CoV-2 ainda não tiveram um grande impacto no curso da pandemia mas podem vir a ter no futuro.
Quando o COVID-19 se espalhou pelo mundo este ano, David Montefiori se perguntou como o vírus mortal por trás da pandemia poderia estar mudando conforme passava de pessoa para pessoa. Montefiori é um virologista que passou grande parte de sua carreira estudando como mutações fortuitas no HIV o ajudam a escapar do sistema imunológico. A mesma coisa pode acontecer com o SARS-CoV-2, pensei.
Em março, Montefiori, que dirige um laboratório de pesquisa de vacinas contra a Aids na Duke University em Durham, Carolina do Norte, contatou Bette Korber, especialista em evolução do HIV e colaboradora de longa data. Korber, um biólogo computacional do Laboratório Nacional de Los Alamos (LANL) em Sante Fe, Novo México, já havia começado a vasculhar milhares de sequências genéticas de coronavírus em busca de mutações que pudessem ter alterado as propriedades do vírus conforme ele percorria o mundo.
Comparado com o HIV, o SARS-CoV-2 muda muito mais lentamente à medida que se espalha. Mas uma mutação se destacou para Korber. Estava no gene que codifica a proteína spike, que ajuda as partículas virais a penetrar nas células. Korber viu a mutação aparecendo repetidamente em amostras de pessoas com COVID-19. Na posição do 614º aminoácido da proteína spike, o aspartato de aminoácido (D, abreviatura bioquímica) estava sendo regularmente substituído por glicina (G) devido a uma falha de cópia que alterou um único nucleotídeo no código de RNA de 29.903 letras do vírus . Os virologistas a chamavam de mutação D614G.
Em abril, Korber, Montefiori e outros alertaram em um pré-impressão postado no servidor bioRxiv que ?o D614G está aumentando em frequência a uma taxa alarmante? 1. Ele rapidamente se tornou a linhagem SARS-CoV-2 dominante na Europa e então se consolidou nos Estados Unidos, Canadá e Austrália. O D614G representava uma ?forma mais transmissível do SARS-CoV-2?, declarou o jornal, que surgiu como um produto da seleção natural.
Essas afirmações desanimaram muitos cientistas. Não estava claro se a linhagem viral D614G era mais transmissível ou se seu aumento indicava algo incomum, disseram eles. Mas o alarme se espalhou rapidamente pela mídia. Embora muitas notícias incluíssem advertências de pesquisadores, algumas manchetes declararam que o vírus estava mudando para se tornar mais perigoso. Em retrospecto, Montefiori diz que ele e seus colegas se arrependem de descrever a ascensão da variante como “alarmante”. A palavra foi apagada da versão revisada por pares do artigo, publicada na Cell em 2 de julho.
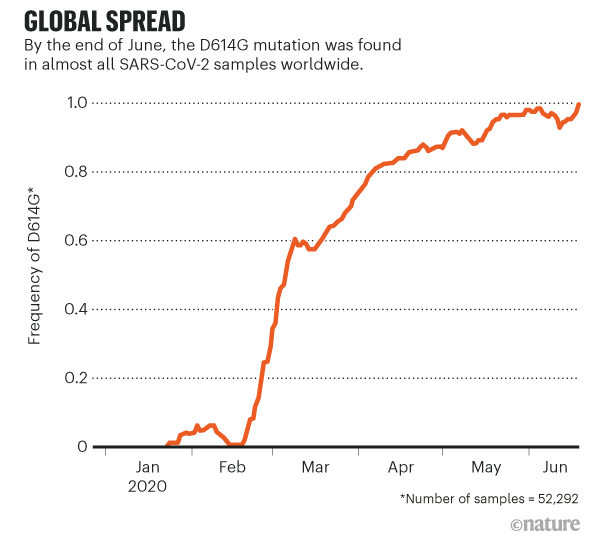
O trabalho despertou um frenesi de interesse no D614G. Mesmo aqueles que estavam céticos de que a mutação tivesse alterado as propriedades do vírus concordaram que era intrigante, por causa de sua ascensão meteórica e onipresença. Por meses, essa linhagem foi encontrada em quase todas as amostras sequenciadas de SARS-CoV-2 (ver ?Propagação global?). ?Essa variante agora é a pandemia. Como resultado, suas propriedades são importantes ?, escreveu Nathan Grubaugh, um epidemiologista viral da Escola de Saúde Pública de Yale em New Haven, Connecticut, e dois colegas em um ensaio Cell sobre as descobertas de Korber e Montefiori.
Até agora, o resultado deste trabalho é menos claro do que a pré-impressão de Montefiori e Korber sugeriu. Alguns experimentos sugerem que os vírus que carregam a variante infectam as células com mais facilidade. Outro trabalho revelou possíveis boas notícias: a variante pode significar que as vacinas podem ter como alvo o SARS-CoV-2 mais facilmente. Mas muitos cientistas dizem que não há provas sólidas de que o D614G tenha um efeito significativo na disseminação do vírus ou que um processo de seleção natural explique seu surgimento. ?O júri está decidido?, disse Timothy Sheahan, coronavirologista da Universidade da Carolina do Norte em Chapel Hill. “Essa mutação pode significar alguma coisa ou não.”
Os pesquisadores ainda têm mais perguntas do que respostas sobre as mutações do coronavírus, e ninguém ainda encontrou qualquer alteração no SARS-CoV-2 que possa levantar questões de saúde pública, afirmam Sheahan, Grubaugh e outros. Mas estudar mutações em detalhes pode ser importante para controlar a pandemia. Também pode ajudar a prevenir as mutações mais preocupantes: aquelas que podem ajudar o vírus a escapar do sistema imunológico, vacinas ou terapias com anticorpos.
Mudança lenta
Logo depois que o SARS-CoV-2 foi detectado na China, os pesquisadores começaram a analisar amostras virais e postar os códigos genéticos online. Mutações – a maioria delas alterações de uma única letra entre vírus de diferentes pessoas – permitiram aos pesquisadores rastrear a propagação ligando vírus intimamente relacionados e estimar quando o SARS-CoV-2 começou a infectar humanos.
Os vírus que codificam seu genoma em RNA, como SARS-CoV-2, HIV e influenza, tendem a pegar mutações rapidamente quando são copiados dentro de seus hospedeiros, porque as enzimas que copiam RNA estão sujeitas a cometer erros. Depois que o vírus da síndrome respiratória aguda grave (SARS) começou a circular em humanos, por exemplo, desenvolveu um tipo de mutação chamada deleção que pode ter retardado sua disseminação4.
Mas os dados de sequenciamento sugerem que os coronavírus mudam mais lentamente do que a maioria dos outros vírus de RNA, provavelmente por causa de uma enzima de “revisão” que corrige erros de cópia potencialmente fatais. Um vírus SARS-CoV-2 típico acumula apenas duas mutações de uma única letra por mês em seu genoma – uma taxa de mudança cerca da metade da influenza e um quarto da do HIV, diz Emma Hodcroft, epidemiologista molecular da Universidade de Basel , Suíça.
Outros dados do genoma enfatizaram essa estabilidade – mais de 90.000 isolados foram sequenciados e tornados públicos (ver www.gisaid.org). Dois vírus SARS-CoV-2 coletados em qualquer lugar do mundo diferem em uma média de apenas 10 letras de RNA de 29.903, diz Lucy Van Dorp, geneticista computacional da University College London, que está rastreando as diferenças em busca de sinais de que eles conferem um vantagem evolutiva.
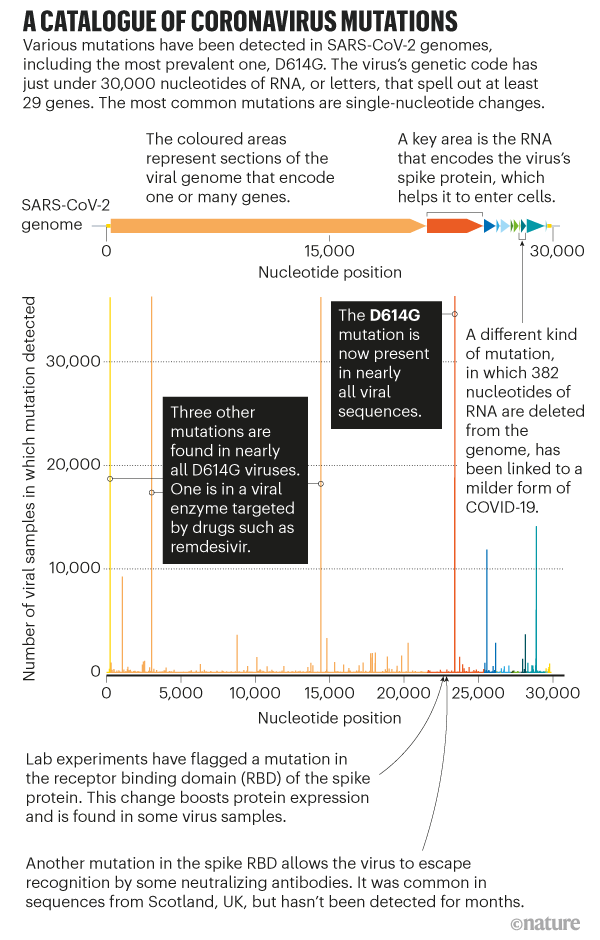
Apesar da lenta taxa de mutação do vírus, os pesquisadores catalogaram mais de 12.000 mutações nos genomas do SARS-CoV-2. Mas os cientistas podem detectar as mutações mais rápido do que conseguem entendê-las. Muitas mutações não terão consequências para a capacidade do vírus de se espalhar ou causar doenças, porque não alteram a forma de uma proteína, enquanto as mutações que alteram as proteínas têm mais probabilidade de prejudicar o vírus do que melhorá-lo (consulte ‘Um catálogo de mutações de coronavírus ‘). ?É muito mais fácil quebrar algo do que consertar?, diz Hodcroft, que faz parte do Nextstrain, um esforço para analisar os genomas do SARS-CoV-2 em tempo real.
Muitos pesquisadores suspeitam que, se uma mutação ajudou o vírus a se espalhar mais rápido, provavelmente aconteceu mais cedo, quando o vírus saltou para os humanos ou adquiriu a capacidade de se mover com eficiência de uma pessoa para outra. Em uma época em que quase todas as pessoas no planeta são suscetíveis, é provável que haja pouca pressão evolutiva sobre o vírus para se espalhar melhor, então mesmo as mutações potencialmente benéficas podem não florescer. ?No que diz respeito ao vírus, cada pessoa que se trata é um bom pedaço de carne?, diz William Hanage, epidemiologista da Escola de Saúde Pública Harvard T. H. Chan em Boston, Massachusetts. “Não há seleção para fazer melhor.”
Distribuição mais rápida?
Quando Korber viu a rápida disseminação do D614G, ela pensou que poderia ter encontrado um exemplo de seleção natural significativa. A mutação chamou sua atenção por causa de sua posição na proteína spike, que é um alvo importante para anticorpos “neutralizantes” que se ligam ao vírus e o tornam não infeccioso. E os vírus com a mutação também estavam crescendo em frequência em mais de uma parte do mundo.
O D614G foi detectado pela primeira vez em vírus coletados na China e na Alemanha no final de janeiro; a maioria dos cientistas suspeita que a mutação tenha surgido na China. Agora é quase sempre acompanhado por três mutações em outras partes do genoma SARS-CoV-2 – possível evidência de que a maioria dos vírus D614G compartilham um ancestral comum.
O rápido crescimento do D614G na Europa chamou a atenção de Korber. Antes de março – quando grande parte do continente entrou em bloqueio – os vírus ‘D’ não mutados e os vírus ‘G’ mutados estavam presentes, com os vírus D prevalentes na maioria dos países da Europa Ocidental que os geneticistas fizeram uma amostra na época. Em março, os vírus G aumentaram de frequência em todo o continente e, em abril, eram dominantes, relataram Korber, Montefiori e sua equipe1,2.

Mas a seleção natural em favor dos vírus G não é a única, nem mesmo a mais provável, explicação para esse padrão. O domínio europeu das variantes G pode ser simplesmente aleatório – se, por exemplo, a mutação for um pouco mais comum nos vírus que chegaram à Europa. Um pequeno número de indivíduos parece ser responsável pela maior parte da propagação do vírus, e uma inclinação inicial e casual em favor dos vírus G poderia explicar a aparente aquisição da linhagem agora. Esses “efeitos fundadores” são comuns em vírus, especialmente quando se espalham sem controle, como o SARS-CoV-2 fez em grande parte da Europa até meados de março.
Korber e seus colegas tentaram descartar um efeito fundador, mostrando em seu preprint1 de abril que o D614G alcançou o domínio rapidamente no Canadá, Austrália e partes dos Estados Unidos (uma exceção foi a Islândia, onde os vírus G presentes no início de seu surto foram superados por vírus D). Analisando os dados de hospitalização de Sheffield, no Reino Unido, a equipe não encontrou evidências de que os vírus portadores da mutação tornassem as pessoas ainda mais doentes. Mas aqueles infectados com vírus G pareciam ter níveis ligeiramente mais elevados de RNA viral em seus narizes e bocas do que aqueles com vírus D.
Muitos cientistas não estavam convencidos de que a ascensão do D614G foi notável – ou tão relevante para a pandemia. ?Achei que a pré-impressão era incrivelmente prematura?, disse Sheahan.
Montefiori diz que a perspectiva dele e de Korber sobre o D614G foi moldada por seu trabalho com o HIV, que descobriu que mesmo mutações aparentemente insignificantes podem ter um efeito profundo em como o sistema imunológico reconhece esse vírus. ?Ficamos alarmados com isso e precisamos ver se está tendo efeito sobre as vacinas?, diz ele.
Corrida de estudos de laboratório
Para examinar mais a fundo se o D614G tornou o vírus mais transmissível, Montefiori avaliou seus efeitos em condições de laboratório. Ele não pôde estudar o vírus natural SARS-CoV-2 em seu laboratório, por causa da contenção de biossegurança necessária. Portanto, estudei uma forma geneticamente modificada de HIV que usava a proteína spike SARS-CoV-2 para infectar células. Essas partículas de ‘pseudovírus’ são um burro de carga dos laboratórios de virologia: elas permitem o estudo seguro de patógenos mortais, como o vírus Ebola, e tornam mais fácil testar os efeitos das mutações.

A primeira equipe a relatar experimentos de pseudovírus no D614G, em junho, foi liderada por Hyeryun Choe e Michael Farzan, virologistas do Scripps Research Institute em La Jolla, Califórnia5. Várias outras equipes postaram estudos semelhantes sobre bioRxiv (experimentos de Montefiori, e os de outro colaborador, apareceram no Cell paper2). As equipes usaram diferentes sistemas de pseudovírus e os testaram em vários tipos de células, mas os experimentos apontaram para a mesma conclusão: os vírus que carregam a mutação G infectaram as células com muito mais habilidade do que os vírus D – até dez vezes mais eficientemente, em alguns casos.
Em testes de laboratório, ?todos nós concordamos que D a G está tornando as partículas mais infecciosas?, diz Jeremy Luban, virologista da Escola de Medicina da Universidade de Massachusetts em Worcester. Mas esses estudos vêm com muitas advertências – e sua relevância para as infecções humanas não é clara. ?O que é irritante são as pessoas vendo seus resultados em ambientes muito controlados e dizendo que isso significa algo para a pandemia. Isso, estamos muito longe de saber ?, diz Grubaugh. Os pseudovírus carregam apenas a proteína spike do coronavírus, na maioria dos casos, e então os experimentos medem apenas a capacidade dessas partículas de entrar nas células, não aspectos de seus efeitos dentro das células, muito menos em um organismo. Eles também não possuem as outras três mutações que quase todos os vírus D614G carregam. ?O ponto principal é que eles não são o vírus?, diz Luban.
Alguns laboratórios estão trabalhando agora com vírus infecciosos SARS-CoV-2 que diferem apenas por um único aminoácido. Eles são testados em culturas de laboratório de células de pulmão humano e das vias aéreas, e em animais de laboratório, como furões e hamsters. Para laboratórios com experiência e recursos de biossegurança para manipular vírus, ?isso é como um trabalho comum?, diz Sheahan. O primeiro desses estudos, liderado por pesquisadores da University of Texas Medical Branch em Galveston, foi relatado em uma publicação preliminar de 2 de setembro6. Ele descobriu que os vírus com a mutação eram mais infecciosos do que os vírus D em uma linha de células de pulmão humano e em tecidos das vias aéreas, e que os vírus mutados estavam presentes em níveis maiores nas vias aéreas superiores de hamsters infectados6.
Mesmo esses experimentos podem não oferecer clareza absoluta. Alguns estudos mostram que certas mutações na proteína do pico no vírus da síndrome respiratória do Oriente Médio (MERS) podem causar doenças mais graves em camundongos – ainda outras mutações na proteína mostram muito pouco efeito em pessoas ou camelos, o provável reservatório para humanos Infecções por MERS, diz Stanley Perlman, coronavirologista da Universidade de Iowa em Iowa City.
O sinal mais claro de que o D614G tem um efeito sobre a disseminação do SARS-CoV-2 em humanos vem de um esforço ambicioso do Reino Unido chamado COVID-19 Genomics UK Consortium, que analisou genomas de cerca de 25.000 amostras virais. A partir desses dados, os pesquisadores identificaram mais de 1.300 casos em que um vírus entrou no Reino Unido e se espalhou, incluindo exemplos de vírus do tipo D e G.
Uma equipe liderada por Andrew Rambaut, um biólogo evolucionário da Universidade de Edimburgo, Reino Unido, o epidemiologista Erik Volz, do Imperial College London, e o biólogo Thomas Connor da Universidade de Cardiff, estudou a disseminação de 62 clusters COVID-19 no Reino Unido semeados por vírus D e 245 por G vírus7. Os pesquisadores não encontraram diferenças clínicas em pessoas infectadas com qualquer um dos vírus. No entanto, os vírus G tendiam a se transmitir um pouco mais rápido do que as linhagens que não carregavam a mudança e formaram grupos maiores de infecções. Suas estimativas da diferença nas taxas de transmissão giram em torno de 20%, diz Volz, mas o valor real pode ser um pouco maior ou menor. ?Não há um grande efeito em termos absolutos?, diz Rambaut.
É possível que o D614G seja uma adaptação que ajuda o vírus a infectar células ou competir com vírus que não carregam a mudança, enquanto altera pouco sobre como o SARS-CoV-2 se espalha entre as pessoas ou através de uma população, diz Rambaut. ?Esta pode ser uma adaptação genuína para humanos ou algumas células humanas?, Grubaugh concorda, ?mas isso não significa que nada mude. Uma adaptação não precisa torná-lo mais transmissível. ”
Grubaugh acha que o D614G recebeu muita atenção dos cientistas, em parte por causa dos artigos de alto perfil que recebeu. ?Os cientistas têm um fascínio louco por essas mutações?, diz ele. Mas ele também vê o D614G como uma forma de aprender sobre um vírus que não tem muita diversidade genética. ?O virologista em mim olha para essas coisas e diz que seria muito divertido estudar?, diz ele. “Isso cria toda essa toca de coelho com diferentes coisas em que você pode entrar.”
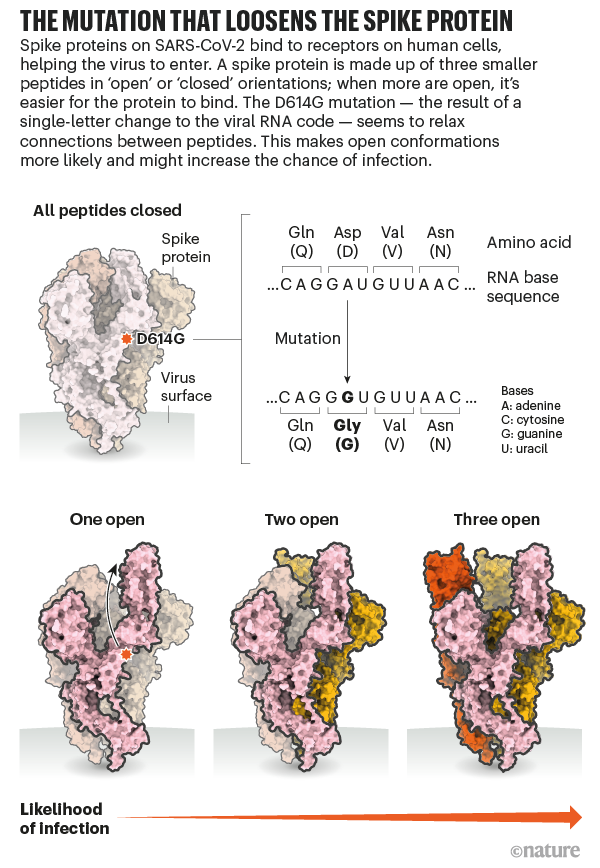
Ele terá companhia. O estudo intenso do D614G deve ajudar a explicar como o SARS-CoV-2 se funde com as células, diz Luban – um processo que pode ser bloqueado por drogas ou direcionado por uma vacina. Em uma versão atualizada de seus experimentos com pseudovírus publicados no bioRxiv em 16 de julho de 2008, a equipe de Luban usou microscopia crioeletrônica para analisar a estrutura das proteínas de pico com a alteração D614G. A proteína spike é composta por três peptídeos idênticos em uma orientação “aberta” ou “fechada”. Pesquisas anteriores sugeriram que pelo menos dois dos três peptídeos precisam ser abertos para que a partícula viral se funda com a membrana celular9, e a equipe de Luban descobriu que os vírus que carregam a variante do pico G tinham muito mais probabilidade de estar neste estado (ver ‘ A mutação que solta a proteína spike ‘). O trabalho de modelagem computacional de Montefiori e Korber, liderado pelo colega de Korber em LANL, Sandrasegaram Gnanakaran, chegou à mesma conclusão10. ?Parece que esta máquina molecular está preparada para funcionar de uma maneira que D não está?, diz Luban.
Sem fuga de anticorpos – ainda
A maioria das evidências disponíveis sugere que o D614G não impede que os anticorpos neutralizantes do sistema imunológico reconheçam o SARS-CoV-2, como Montefiori havia se preocupado. Isso pode ser porque a mutação não está no domínio de ligação ao receptor da proteína spike (RBD), uma região que muitos anticorpos neutralizantes têm como alvo: o RBD se liga à proteína celular-receptora ACE2, uma etapa chave na entrada do vírus nas células.
Mas estão surgindo evidências de que outras mutações podem ajudar o vírus a evitar alguns anticorpos. Uma equipe liderada pelos virologistas Theodora Hatziioannou e Paul Bieniasz, da Universidade Rockefeller em Nova York, modificou geneticamente o vírus da estomatite vesicular – um patógeno de gado – para que usasse a proteína spike SARS-CoV-2 para infectar células e o cultivou em a presença de anticorpos neutralizantes. Seu objetivo era selecionar mutações que permitissem que a proteína spike evitasse o reconhecimento de anticorpos. O experimento gerou mutantes de proteína de pico que eram resistentes a anticorpos retirados do sangue de pessoas que se recuperaram de COVID-19, bem como a potentes anticorpos “monoclonais” que estão sendo desenvolvidos em terapias. Cada uma das mutações de pico foi encontrada em sequências de vírus isoladas de pacientes, relatam Hatziioannou, Bieniasz e sua equipe – embora em frequências muito baixas que sugerem que a seleção positiva ainda não está tornando as mutações mais comuns11.
Outros cientistas estão tentando ficar à frente da evolução do SARS-CoV-2, prevendo quais mutações são provavelmente importantes. Jesse Bloom, um virologista evolucionário do Fred Hutchinson Cancer Research Center em Seattle, Washington, liderou uma equipe que criou quase 4.000 versões mutadas do RBD da proteína spike e mediu como as alterações afetaram a expressão da proteína spike e sua capacidade de se ligar para ACE2. A maioria das mutações não afetou ou prejudicou essas propriedades, embora algumas as tenham melhorado12. Algumas dessas mutações foram identificadas em pessoas com COVID-19, mas a equipe de Bloom não encontrou sinais de seleção natural para nenhuma das variantes. ?Provavelmente, o vírus se liga ao ACE2 tão bem quanto precisa agora?, diz ele.
Os pesquisadores não testaram se alguma das mutações permite ao vírus impedir a ação dos anticorpos, mas os resultados de sua equipe sugerem que tais mudanças são possíveis. ?É uma possibilidade, mas não uma certeza, de que o vírus adquira mutações que alteram sua suscetibilidade a anticorpos e imunidade?, diz Bloom.
Com base na experiência com outros coronavírus, isso pode levar anos. Estudos de coronavírus do resfriado comum, coletados em várias estações, identificaram alguns sinais de evolução em resposta à imunidade. Mas o ritmo da mudança é lento, diz Volker Thiel, virologista de RNA do Instituto de Virologia e Imunologia de Berna. “Essas cepas permanecem constantes, mais ou menos.”
Com a maior parte do mundo ainda suscetível ao SARS-CoV-2, é improvável que a imunidade seja atualmente um fator importante na evolução do vírus. Mas à medida que a imunidade da população aumenta, seja por infecção ou vacinação, um fluxo constante de mutações evasivas do sistema imunológico pode ajudar a SARS-CoV-2 a se estabelecer de forma permanente, diz Sheahan, podendo causar sintomas leves quando infecta indivíduos com algum resíduo imunidade de uma infecção anterior ou vacinação. “Eu não ficaria surpreso se este vírus fosse mantido como um coronavírus causador de resfriado mais comum.” Mas também é possível que nossas respostas imunológicas a infecções por coronavírus, incluindo a SARS-CoV-2, não sejam fortes ou duradouras o suficiente para gerar pressão de seleção que leva a cepas de vírus significativamente alteradas.
Mutações preocupantes também podem se tornar mais comuns se as terapias com anticorpos não forem usadas com sabedoria – se as pessoas com COVID-19 receberem um anticorpo, que pode ser impedido por uma única mutação viral, por exemplo. Coquetéis de anticorpos monoclonais, cada um dos quais reconhecendo várias regiões da proteína spike, podem diminuir as chances de que tal mutação seja favorecida pela seleção natural, dizem os pesquisadores. As vacinas causam menos preocupação nesse aspecto porque, como a resposta imune natural do corpo, elas tendem a desencadear uma variedade de anticorpos.
É até possível que a mudança no D614G torne o vírus um alvo mais fácil para vacinas, descobriu a equipe de Montefiori em um estudo publicado no bioRxiv em julho de 13. Ratos, macacos e humanos que receberam uma das várias vacinas experimentais de RNA, incluindo uma que está sendo desenvolvida pela fabricante de medicamentos Pfizer na cidade de Nova York, produziram anticorpos que se mostraram mais potentes no bloqueio dos vírus G do que os vírus D.
Com os vírus G agora onipresentes, a descoberta é uma ?boa notícia?, diz Montefiori. Mas, como um cientista que viu o HIV sofrer mutação para evitar muitas vacinas desenvolvidas contra ele, ele permanece desconfiado do potencial do SARS-CoV-2 de escapar das respostas da humanidade. Luban concorda: “Precisamos manter nossos olhos abertos para mudanças adicionais.”
Publicado em 08/09/2020 22h18
Artigo original:
Estudo original:
Achou importante? Compartilhe!
Assine nossa newsletter e fique informado sobre Astrofísica, Biofísica, Geofísica e outras áreas. Preencha seu e-mail no espaço abaixo e clique em “OK”:




